RESEARCH
不規則な収納が生む自由
私たちヒトの体は約60兆個の細胞からなり、その細胞1つ1つに全長2メートルのDNAが入っています。こんなに長いDNAが、どのように収納され使われているのでしょうか。細胞が分裂する際にできる染色体のDNAはヒストンの芯に巻き付いた直径11nm(ナノメートル=1mの10億分の1)のヌクレオソーム構造をとっています。それが規則正しく折り畳まれて直径30nmのクロマチン線維となり、さらに集まるという階層構造を形成していると考えられてきました。ところが、国立遺伝学研究所の前島一博さんが詳細に調べたところ、定説のようなクロマチン線維は存在せず、より柔軟でダイナミックな姿が浮かび上がってきました。教科書に書かれてきた図は違うのではないか。こんな基本を問う研究です。
1.これまで考えられていた染色体構造
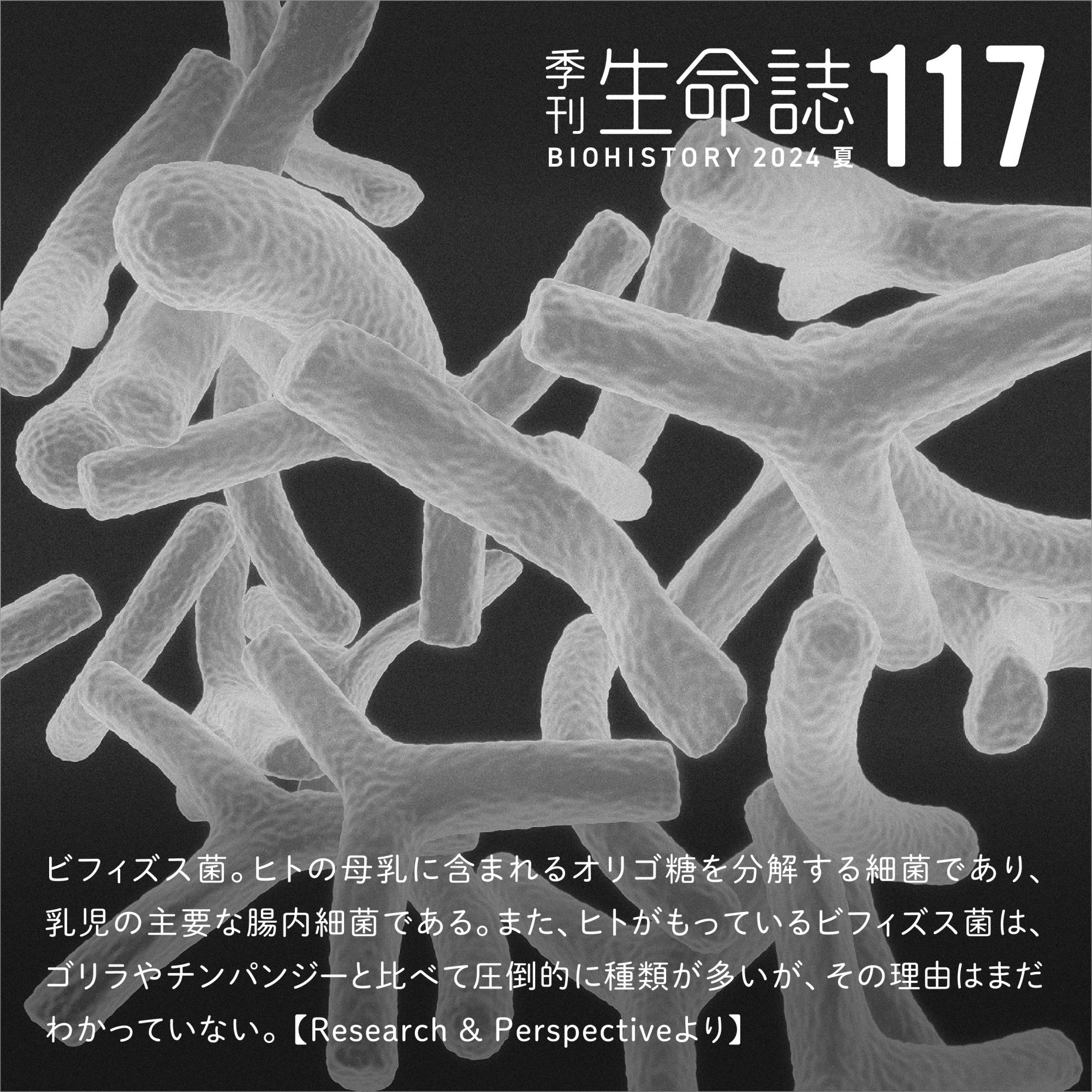
数千万種とも言われる多様な生きものはすべて細胞からなり、そこにあるDNAのもつ情報をもとに活動しているという普遍性を持っている。細胞内のDNAの存在のしかたは、細胞のはたらき方を知る基本である。たとえば、私たちヒトの体は成人で約60兆個の細胞からなり、各細胞に全長約2mにも達するDNAが収められている。細胞は体をつくりあげる時だけでなく、常に分裂しており、その際、DNAが切れたり絡まったりしないように凝縮し、染色体と呼ばれるDNAの束になる(写真1、動画1)。染色体は、19世紀末からその存在が知られている。
(写真1) 細胞分裂(DNAを染色)のようす
DNAは凝縮・複製されて染色体になり、2つの新しい細胞が形成される。
(動画1) 細胞分裂(DNAを染色)のようす
ヒト培養細胞の分裂の様子。赤色に染めているのが染色体、細胞全体を緑で染めている。30分に1度、約30時間にわたって撮影した。
引用文献:Nat Struct Mol Biol.(2010)17:1065-71
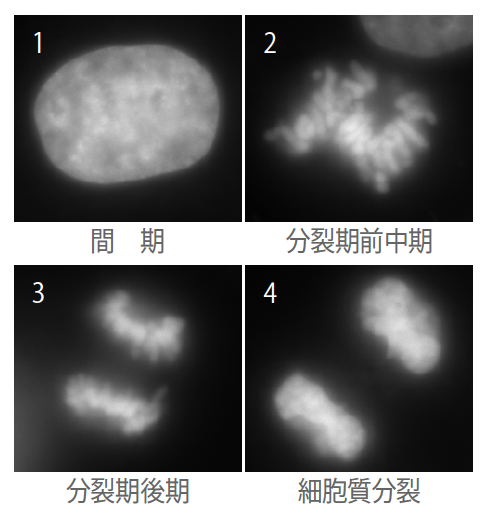
1976年、イギリスのクルーグ(註1)らは、染色体を構成する最小基本単位であるヌクレオソーム線維がらせん状に規則正しく折り畳まれて、直径約30nmのクロマチン線維をつくるという染色体構造モデルを提唱した。現在広く受け入れられている染色体構造は、このクロマチン線維がらせん状に巻かれて100nmの線維をつくり、つぎに200-250nm、さらには500-750nmと、規則正しいらせん状の階層構造(積み木構造)を形成するというものである(図1)。実際、分子生物学の有名な教科書である『細胞の分子生物学』にも高等学校の生物IIの教科書にもこの図が載っている。
(図1) これまで考えられていた染色体の構造
30nmのクロマチン線維が、規則正しいらせん状の階層構造を形成していると考えられていた。
(註1) クルーグ
イギリスの生物物理・構造生物学者で、1982年に「電子顕微鏡による構造解析法の開発と生物学的に重要な核酸ータンパク質複合体の立体構造の解明」によりノーベル化学賞を受賞した。
2.30nmの構造が見えない
私たちは一貫して、ゲノムの本態であるDNAの束ねられ方や収納のされ方に着目して研究を続けてきた。2004年頃からは、ドイツEMBLのグループと共に、生きたままに近い状態の細胞を観察できる電子顕微鏡(クライオ電子顕微鏡)を用いて、ヒト細胞の分裂を解析している。その解析では、ヌクレオソーム線維の存在を示す「直径11nmの構造」は観察できたが、定説のクロマチン線維モデル「直径30nmの構造」は観察できなかったのである(写真2)*1。そこで、クロマチン線維は存在しないのかもしれないと疑い始めた。しかし、クライオ電子顕微鏡にはいくつかの技術上の問題があった。70nm程度の薄い切片を観察するため、染色体のごく一部しか解析できないことや、得られた画像の濃淡が極めて薄いことなどである。このため、この結果だけでは染色体に30nm構造が無いと断定することはできない。そこで私たちは、X線散乱を用いた構造解析をおこなった。

(写真2) クライオ電子顕微鏡撮影の写真
30nmの構造があれば、写真内の右上のようなモデル構造が見られるが、染色体上には見られない。
*1:引用文献
Proc Natl Acad Sci USA (2008) 105: 19732–19737
3.30nm構造の正体
X線散乱とは、タンパク質などが集まった構造体にX線を当てると、その構造体の規則性に応じた散乱パターンが得られる原理を用いた解析方法である。25年以上前に、イギリスのグループがX線散乱による染色体の構造解析をおこなったところ、30nm程度の構造を観察しており、これが染色体に30nmのクロマチン線維が存在する強力な根拠の一つとなっている*2。私たちは、X線散乱を用いて詳細に構造を解析するために、理研・播磨研究所の大型放射光施設SPring8で放射光を用いた。放射光は強力なX線である。その結果、私たちの解析でも、染色体に30nmの構造が観察されたのである。これは、クライオ電子顕微鏡での観察と矛盾する。
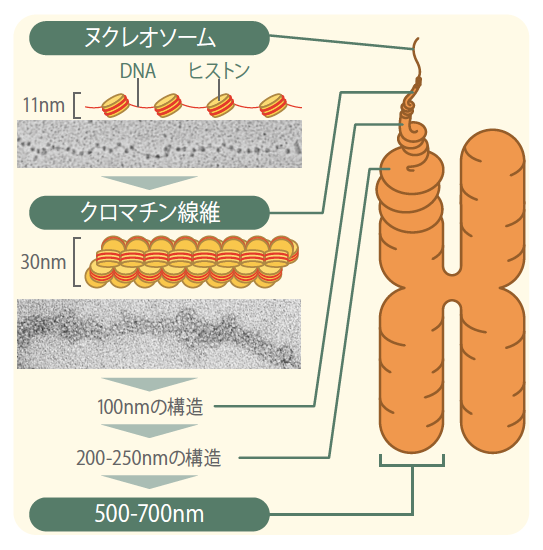
そこで、クライオ電子顕微鏡とX線散乱の観察結果を詳細に検討した。驚いたことに、私たちがX線散乱の解析に用いた分裂期染色体をクライオ電子顕微鏡で観察したところ、染色体表面に多数の黒い粒が付着していることがわかった。後に、この黒い粒は、約30nmの間隔で規則正しく並んだリボソームであることが判明した。このため、X線散乱による30nmの構造の正体は、染色体本体ではなく、その表面に付着したリボソームではないかと予想した。リボソームはタンパク質合成をする場で、細胞内に大量に存在する。また、RNAとタンパク質からなる大きさ20nm以上の巨大な複合体である。さっそく、リボソームを取り除いた染色体をX線解析したところ、今度は30nm構造は観察されなかった。これで、染色体に30nmのクロマチン線維が存在する根拠はなくなったわけである(図2)*3。
(図2) X線散乱で見えた30nm構造はリボソームだった。
リボソームを染色体から取り除くと、30nmの構造を示す値が出なかった。6nmはヌクレオソームの厚さ、11nmはヌクレオソームの直径。
次は、染色体内の階層構造(積み木構造)の有無の確認である。染色体を丸ごと構造解析できるX線散乱装置をつくった西野博士らと共同で染色体照射をおこなった(写真3)。染色体直径に相当する1μm(1mの1000分の1、1nmの1000倍)までの範囲を調べ、詳細に解析したところ、100nm、200-250 nmにあたる線維は観察されず、ヌクレオソーム線維を示す11 nmの構造しか存在しなかった。つまり、30nmクロマチン線維も、それがさらに規則正しく束ねられた高次の構造も存在しないのである。私たちの実験は、染色体にはヌクレオソーム線維が不規則に収納されているという、まったく新しい染色体像を示している*3。
これまで、電子顕微鏡で30nmのクロマチン線維が観察されていたのはどうしてだろう(図1)。試験管内の実験では、ヌクレオソーム線維の濃度がうすく、線維内のとなり同士のヌクレオソームが結合しやすいため、30nmのクロマチン線維を作りやすいことがわかっている。また、通常の電子顕微鏡は試料を真空中にさらすため、化学固定・アルコール脱水・樹脂包埋・切片作成そして染色という複雑なプロセスを経る必要がある。この過程で、人工的に凝集したヌクレオソーム線維を30nmの構造として観察していた可能性も高い。一方、私たちの実験が用いたクライオ電子顕微鏡は「生きている」状態を観ている。細胞を高圧下で急速凍結し、極低温下(-150 度)で薄片にし、それを極低温下でそのまま観察するからである。また、X線散乱でも化学固定・アルコール脱水なしで、溶液のなかの染色体を観察する。そのため、ヌクレオソーム線維の凝集が起きなかったのである。

(写真3) 実験を行った理研・播磨研究所の大型放射光施設SPring8
写真(左):全景
写真(中):実験部屋の外観(BL45XU)
写真(右):X線散乱装置。右側からX線が照射される。
*2:引用文献
Langmore JP, Paulson JR (1983) J Cell Biol 96: 1120–1131など
*3:引用文献
The EMBO Journal(2012)17:1644-53
4.なぜ不規則な収納で染色体の形をつくれるのか
全長2mにもおよぶヒトゲノムDNAが細胞の染色体の中に不規則に収納されていることは、決まった染色体の形から考えると意外に思われる。しかしきっちりと束ねられた構造を作るには大きなエネルギーを必要とするので、最小限の秩序を保つ構造だけを作り、あとは自由に凝縮させて、なるべくエネルギーを使わずに効率良く染色体を作る方が合理的とも考えられる。近年、コンデンシンやトポイソメラーゼIIと呼ばれる特定のタンパク質が軸となることで、ヌクレオソーム線維をゆるやかに束ね、X型の染色体のかたちを構成できることがわかり始めた。(図3中央)
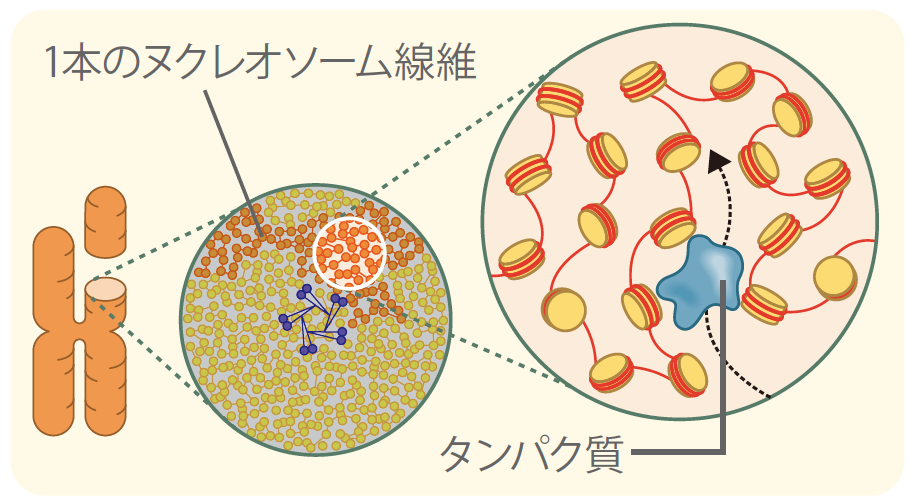
(図3) 新しいDNA収納モデル
ヌクレオソーム線維は分子相互作用により常に揺れているような状態を保ったまま不規則に収納されている。
5.動きやすさは探しやすさにつながる
私たちは、染色体でみられるヌクレオソーム線維の不規則な収納は、染色体になる前の間期の段階からすでに存在しており*4、RNAポリメラーゼ(註2)や転写因子(註3)のDNAへの結合を促進させていると考えている。間期は、核内でDNA情報の検索や転写が盛んに行われている時期である。この時、DNAがある程度の自由度を持って不規則に収納されていれば、個々のヌクレオソームが動ける余地が増え、遺伝情報の検索に効率が良いだろう(図3右)。実際私たちは、このような個々のヌクレオソームの動き(ゆらぎ)を生きた細胞で観察することに成功しており、これが情報検索に重要であるというデータも得られつつある。*5
これまで、各遺伝子の転写開始のしくみは詳細に研究されてきたが、情報検索に関する研究はほとんどなされていない。私たちの興味は、個別の遺伝子ではなく、生きものにおけるゲノムDNAの普遍的な収納や情報検索の大まかなしくみにある。ここから、生きている状態をよりよく知ることができるはずだ。さらに、将来これが新しい概念によるメモリデバイスや情報検索システムの開発につながるのではないかとも考えている。
(註2) RNAポリメラーゼ
遺伝情報はDNAから伝令RNAや運搬RNA等の状態を経てタンパク質が作られる。RNAポリメラーゼはDNAを鋳型に、対応する4つの塩基を順々にくっつけて伝令RNAを合成する複合タンパク質。
(註3) 転写因子
DNAからRNAを合成する段階を転写と呼ぶ。転写因子はDNAと特異的に結合して転写の促進や抑制を行う。
*4:実際にそのデータを得た論文を2012年7月に発表した。
Nucleus. (2012) 3:404-410.
*5:実際にそのデータを得た論文を2012年12月に発表した。
Cell Rep. (2012) 2:1645-56

前島一博(まえしま かずひろ)
1999年大阪大学大学院博士課程修了。博士(医学)。ジュネーヴ大学ポスドク研究員、理化学研究所専任研究員を経て、2009年より国立遺伝学研究所・構造遺伝学研究センター教授。
研究室のWebページはこちらから
https://www.nig.ac.jp/nig/ja/research/organization-top/laboratories/maeshima
![[科学のコンサートホール]BRH JT生命誌研究館](/common/img/logo.svg)