年度別活動報告
年度別活動報告書:2013年度
脊索動物と節足動物の共通祖先を理解する 2−1 オオヒメグモの胚発生を支配する分子メカニズムの解明
小田広樹(主任研究員 秋山-小田康子(特別研究員)
佐々木瑞希(奨励研究員) 野田彰子(研究補助員)
逸見なつき(大阪大学大学院生)
はじめに
多細胞動物は形態や発生様式に基づいて「門」と呼ばれる高次分類群(例えば、ヒトを含む脊索動物門や昆虫を含む節足動物門)に区分されているが、それぞれの分類群を特徴付ける形態が進化してきた過程やその進化の仕組みを理解することは簡単ではない。その大きな要因のひとつは、分類群を特徴づける、機能性の高い進化的に安定な形態であっても、その形態を形作るための発生過程やゲノム情報(遺伝子発現制御機構や遺伝子ネットワークなどを含む)は進化の過程で多様に変化しうることにある。このことは、もし祖先の状態から大きく変容してしまったゲノムを持つ動物種だけを対象として比較解析を進めた場合、生物進化の歴史を誤って再構成してしまう可能性があることを意味する。
例えば、体の軸に沿った反復構造の存在は、ヒトなどの脊椎動物を含む脊索動物門と昆虫やクモを含む節足動物門に共通して見られる特徴であるが、その反復パターンを形作る遺伝的仕組みに両者が同一起源であることを示唆するような共通性は見出されていない。しかし、だからと言ってその類似の形態的特徴が異なる起源から別々に進化したと断言することはできない。なぜならば、共通祖先において存在した反復パターンを形成する仕組みは、発生プログラムの出力となる反復パターンを保持したまま、その形成過程に関わる遺伝子成分や遺伝子ネットワークを現存ゲノムの比較では追跡できないほど大きく変化させて多様化したかもしれないからである。このような可能性を考えた場合、それぞれの動物門の中での遺伝的仕組みの多様性の程度を大局的に把握することが動物門間の進化的関係を理解する上で重要であるが、また同時に、特定の形態やパターンを発生プログラムの出力として出し続けながらそのプログラムがどのように進化し、多様化しうるかを、システム生物学や数理生物学を取り入れて理解することも重要となっている。
私たちはこれまで、独自のモデル生物種として、節足動物門鋏角類オオヒメグモ (Parasteatoda tepidariorum, 旧名Achaearanea tepidariorum) を用いて、節足動物の基本的体制(ボディープラン)に関わる発生メカニズムの多様性を明らかにしてきた。オオヒメグモの有用性は、技術的利便性に加えて、この動物種によって得られる知見の意外性にある。オオヒメグモ胚では、ヘッジホッグシグナルとディーピーピーシグナルを介した細胞間のコミュニケーションがからだの軸を作り出すのに主要な役割を果たしている1) 2)。とりわけ、ヘッジホッグシグナルの初期胚での役割は、節足動物の多様化過程において発生プログラムが大きく変化したことを象徴的に示している。オオヒメグモではヘッジホッグシグナルが胚全体の極性形成を支配しているのに対して、同様の役割をショウジョウバエでは転写因子のビコイドが果たしている3)。この「ヘッジホッグ 対 ビコイド」の違いは、オオヒメグモ初期胚が細胞性であるのに対して、ショウジョウバエ初期胚が多核性であることとも整合性があり、クモとハエ、または鋏角類と昆虫類の比較が、節足動物の形態の保守的進化の中で多様な発生過程がどうして進化できたのかを理解するための絶好の研究機会を提供する。
だが、形態の保存と、発生過程の多様化の関係を理解するには、実験データの蓄積に基づく現存動物種間の比較だけでは十分ではない。「一方向に流れる時間」との関係を明らかにするために、数理モデリングなどの数学的手法を持ち込むことも必要不可欠である。実際に、一部でこのような試みは始まっている4)。私たちも、クモ初期胚を素材として、この方向の研究を推進したいと考えており、本年度、次世代シーケンサーを利用した初期胚での遺伝子発現の網羅的定量解析や、縞パターン形成の定量的解析、上皮細胞シートのコンピュータ・モデリングなどに着手した。本年度得られた結果は、その数理解析に向けてオオヒメグモ胚の可能性の高さを示すものであった。
結果と考察
(1)クモ初期胚でヘッジホッグシグナルに制御される遺伝子のゲノムワイドな探索
私たちはこれまでの研究で、ヘッジホッグシグナルがオオヒメグモ初期胚において胚全体の極性形成を支配することを明らかにした2)。ヘッジホッグシグナルが制御する遺伝子群を網羅的に探索することは、クモ胚の極性形成や体軸形成に関わる遺伝子のネットワーク全貌の解明につながると考え、ヘッジホッグ遺伝子に対するparental RNA interference (pRNAi)と昨年度導入されたイルミナMiSeqを利用したRNA sequencing (RNA-seq)を組み合わせた解析を開始した。ネットワークの解明が進めば、他の動物の情報と比較することにより、体軸形成の進化やヘッジホッグシグナルの役割の進化に対する理解が進むことも期待できる。
pRNAiは目的とする遺伝子(標的遺伝子)に対する2本鎖RNAを母グモに注射することで胚における標的遺伝子の発現を抑制する方法であり、pRNAi胚とコントロール胚で遺伝子発現を比較することにより、標的遺伝子の抑制に起因して発現変動する遺伝子を同定しうることを既に示している5)。今回もこの方法を利用し、1匹のメスが産む卵を対応あるグループとして、ヘッジホッグに対する二本鎖RNAの注入前と、注入後約15日(pRNAiの効果が中程度)、約20日(pRNAiの効果が最大)に産まれた卵が初期ステージ3、後期ステージ5になった時点でmRNAを抽出し、合計2グループのRNA-seqライブラリーを作製した。ライブラリー作製の際にサンプルに産卵日を識別する3種類のインデックス配列を持たせることで、同時に3サンプルのシーケンスを可能とした。現在シーケンスを進めているところであるが、これまでの試行ではMiSeq1回のランでそれぞれのインデックスあたり600万〜900万リードを得ることができた。1回のランで得た配列情報を、昨年度のロシュの次世代シーケンサーによる解析で得られたcDNA配列情報をリファレンスとしてマッピングし、マップされたリードをカウントすることでサンプルごとの発現量の違いを定量した。ヘッジホッグ遺伝子自身と、ヘッジホッグシグナルにより制御されていることが既に分かっている遺伝子(otd, opa, ptc)の発現が確かに落ちていることが確認でき(図1)、この方法が有効であることが示唆された。今後は現在解析中のオオヒメグモゲノム情報をリファレンスとして用いるための準備を行う。また、数百万〜数千万のリードを扱うための統計的解析手法に関しても検討を進める。さらに、ヘッジホッグシグナルの負の制御因子としてはたらくパッチトのpRNAi胚を用いて同様のRNA-seq解析を行い、ヘッジホッグシグナルが活性化している状態での遺伝子発現に関しても解析を進める計画である。
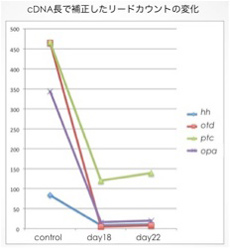
図1.ヘッジホッグpRNAi胚のRNA-seq解析の予備的結果
(2)クモ初期胚における3つの異なる縞パターン形成様式の定量的特徴付け
動物の体節形成は遺伝子発現の空間的周期パターン(周期的縞パターン)の形成を伴う。この縞パターン形成は、発生生物学と数理生物学の両方面からのアプローチが可能な研究対象であるだけでなく、細胞運動や細胞分裂を扱う細胞生物学や発生プロセスの多様化を扱う進化生物学とも結びつき、奥行きの深い研究対象である。私たちは、これまでのクモ胚の解析から、このクモの縞パターン形成現象には、数理生物学を始め、多方面の研究分野の興味を集める要素が詰まっていると考えており、この主張を具体的な形で示すために、縞パターン形成プロセスの定量的解析を行った。定量的発現解析の対象とした遺伝子はヘッジホッグ遺伝子と、ヘッジホッグのレセプターをコードしているパッチト遺伝子であり、両者はこれまでの研究で、少なくとも予定頭部と予定後体部で縞パターン形成の最も初期の段階から発現が始まり、以後発現が継続することが分かっている。以下では、簡略化のために、ヘッジホッグの解析を述べる。
オオヒメグモ一匹の成熟雌個体が一度に200個以上の卵を産み、それらがかなり同調性の高い発生を示すことを利用して時空間パターンの変化を統計的に再構成した。まず、ひとつの卵嚢由来の胚を少数ずつ、20時間にわたって2時間の間隔で固定して別々にプールし、それらを使って蛍光in situハイブリダイゼーションで染色した。染色した胚は共焦点レーザー顕微鏡で観察し、三次元画像データを取得した後、画像解析ソフトImarisとImageJを用いて、胚の表面細胞層内の前後軸に沿ったシグナル強度を取得した。11の時間群(0時間から2時間おきに20時間まで)からそれぞれ約10個ずつ、合計107個の胚を解析し、シグナル強度の計測値を得た。同時に、胚発生の進行度合いを表す指標として、胚領域の前後方向の長さ (Lap) と頭部-胸部境界に沿った背腹方向の長さ (Ldv) の比率 (Lap/Ldv、前後-背腹比) を計測した。Lap/Ldvの対数値を横軸にとって、すべての胚のシグナル強度のプロファイルを一つのグラフに表した(図2; 頭部と胸部の境界を便宜的に前後軸座標の原点とした)。また、胚固定のタイミングと前後-背腹比の分布変化を対応づけることにより、発生時間と胚形の関係を見積もった。
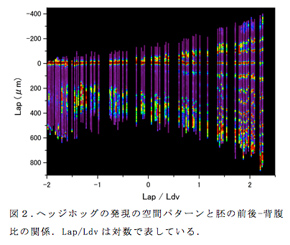
このデータセットを使用して、周期的縞パターンの形成につながるヘッジホッグの発現のダイナミクスを体の領域ごとに調べた。その結果、予定頭部領域では私たちの以前の研究で示したように5)、既存の縞が連続する2回の縞分裂を受けて3つの縞が形成される過程が再構成された。今回の定量的解析で新たに、波のピークの分裂に先行して波の形状が前後方向に非対称になっていることが明らかになった。そしてさらに、上皮組織の収斂伸長に伴って、波の前後幅が40 µm程度(6細胞分に相当)に広がったときに波のピークを2つに分ける谷が表れ始めることが分かった。
予定胸部領域と予定後体部領域では、頭部領域で見られる縞分裂とは異なる様式で周期縞パターンが形成されていることが考えられるが、定量的な解析はまだ不完全である。今後縞パターン形成のメカニズムを、数学的手法を用いて解析することによって、細胞集合体の中で起こる縞パターン形成を説明する理論基盤の構築に貢献したい。オオヒメグモは、比較的均一な細胞からなる一続きのパターン形成場の中に、ひとつのゲノムによって支配される3つの異なる縞パターン形成様式が現れていることから、縞形成能力をもつ多細胞体システムの発生、進化、多様化の研究を実験と理論の両面から遂行できる優れたモデルになりうると考えている。
(3)ヘッジホッグ発現波のダイナミクスを制御するメカニズムの解析
オオヒメグモのステージ5において胚盤の縁に沿って1〜2列の細胞に出現するヘッジホッグの縞状の発現が、ステージ6の間にダイナミックに後方に向かって細胞から細胞へと伝播し、その後、この遺伝子発現波は(2)で示したように2回繰り返し前後に分裂する。私たちは以前の研究で、ヘッジホッグシグナルがオルソデンティクルとオッドペアードの発現を支配する一方、これらの遺伝子活性によってヘッジホッグの発現がフィードバック制御を受けていることを示した5)。そして昨年度までの研究で、ウイングレスシグナル経路の構成因子であるアルマジロとアキシンもそのヘッジホッグの発現ダイナミクスに関わることを突き止めた。今年度、このヘッジホッグ発現波の伝播・分裂を制御するメカニズムをさらに深く追究するために、正常胚とアルマジロ胚性RNAi (eRNAi) 胚を多重色蛍光in situハイブリダイゼーションで詳細に調べた。
その結果、2点の注目すべき新しい発見があった。ひとつは、ヘッジホッグ発現波の分裂直前にプレ分裂期と呼ぶべき時期が存在していることである。ヘッジホッグ発現波は始め全体にわたってオルソデンティクルポジティブであるが、分裂の直前になると、発現波の後方部分のオルソデンティクルの発現がなくなるか、または大きく低下し、同時に、ピークをこの後方部分に残したまま、発現波の前後幅をそのピークに対して前方へ広げていく。この発見は(2)の定量的解析の結果とも一致する。この発見の重要な点は、以前のオルソデンティクルeRNAi細胞クローンの解析から、伝播中のヘッジホッグの発現がオルソデンティクル依存的であることが分かっているのに、どうして後方部のヘッジホッグの発現はオルソデンティクルネガティブになっても強い発現を続けられるのかを説明する必要が有ることを示している点である。2つ目の発見は、アルマジロeRNAi細胞クローンでは、オルソデンティクルネガティブになっていたにも関わらず、その細胞クローンの前端部にヘッジホッグの発現波が侵入し、そこでスタティックに(細胞から細胞への伝播をせずに)発現を続けていたことである。この表現型は明らかにオルソデンティクルeRNAi細胞クローンの表現型とは異なっている。これらの発見を合わせると、ヘッジホッグ発現波の伝播から分裂への移行に際し、ヘッジホッグの発現がオルソデンティクルに依存した状態から依存しない状態への転換が発現波の後方部で起こっている可能性が示唆された。そして、その状態転換がアルマジロ活性またはウイングレスシグナルに対抗する因子で制御されている可能性がある。今後は、オッドペアードやアキシンの解析も加えて、これらの可能性を調査したい。また、地道な機能スクリーニングを通して、この現象に関わる新たな因子の同定も進めていきたい。
(4)細胞再配列を実現する上皮細胞シートのモデリング
オオヒメグモ胚では(おそらく、その他のクモ胚でも)、確率的要素を多く含む細胞再配列と細胞分裂を伴ってパターン形成(例えば、背腹パターンや体節パターンの形成)が進行する。この点において、クモの胚発生は、モザイク卵としてよく研究に使われてきたホヤや線虫の胚発生と対照をなす。クモの胚発生を観察すると、個々(細胞レベル)の乱雑性と全体(組織レベル)の秩序を結びつけるものは何か?という基本的な問題を考えることの必要性に迫られる。この問題に取り組むには、それぞれの細胞が有する①遺伝情報(遺伝子ネットワーク)と、細胞と細胞の間で繰り広げられる②力学的相互作用、そして③化学的相互作用を一つの枠組みで考える必要があるであろう。これら3つの要素すべてを兼ね備えた数理モデルの構築ができれば、多細胞集合体の発生と進化の基本原理を探究するための具体的な方法論を得ることになると私たちは考えている。クモ初期胚は、数学表現と相性のよい形態と細胞構成を持つこと、細胞間相互作用に大きく依存した発生メカニズムを有することなどから、数理モデルの素材として大きな可能性をもつ。私たちは、この方向の研究を推進するための第一歩として、オオヒメグモ初期胚の胚盤上皮を模した二次元上皮細胞シートのコンピュータ・モデリングを試みた。
プログラミングにはオブジェクト指向言語のjavaを使用した。この言語を用いた理由は主に以下の4つである。
1)オブジェクト指向プログラミングが生物の仕組み(階層性や構成オブジェクトの独立性など)と相性がよい。2)グラフィクスが手軽に使用できる。3)汎用性や拡張性に優れる。4)オペレーティング・システムに依存せず、計算スピードが十分に速く、無料で使える。
各上皮細胞を多角形とみなして、アドへレンスジャンクションと細胞骨格の働きに起因する力学的エネルギーを反映した関数を定義した上で、そのエネルギー値を極小にするように細胞の頂点の位置を時間ごとに微小距離移動させた。また、隣同士の頂点がある距離以内に近づいたら、その頂点を結ぶ辺を隣の細胞との間で組換えて、細胞の再配列が起こるようにした。このような上皮細胞モデリングはこれまでに多数の報告6) 7)があるが、私たちはそれらを参考にしつつ、いくつかの点で独自のアルゴリズムを組んだ。最も大きな違いは、乱数の組を発生させてそれぞれの細胞の頂点を動かすことにした点である。乱雑性と秩序・再現性の関係を解析するための工夫のひとつである。細胞の再配列が細胞内で作用する張力を操作するだけで起こるかを試すために、一部の細胞で他の細胞と条件を変えてシミュレーションを行ってみたところ、細胞の再配列が観察された(図3)。今後は、細胞分裂の導入、拡散因子の導入、遺伝子ネットワークの導入を順次進めていきたい。
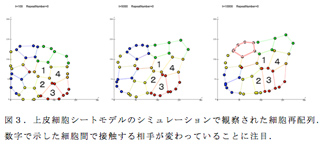
(5)オオヒメグモDNA塩基配列データベースの公開
私たちが有用性を見出したオオヒメグモは、欧米を中心に使う研究者が増えており、新しいモデル生物として世界に受け入れられている。国際的ゲノム配列決定プロジェクトi5k (insect and other arthropod genome sequencing initiative) でも、オオヒメグモゲノムの塩基配列決定が進められている。私たちは研究で得られたオオヒメグモの遺伝子の塩基配列を公共のDNAデータバンクに登録してきたが、それらの情報は他の生物種の情報の中に埋もれてしまい、クモに興味を持つ研究者にとって利用しやすい形で情報が提供されているわけではない。そこで私たちは、オオヒメグモの研究への利用を世界レベルで促進するために、これまでの私たちのEST解析で得られた配列情報をオープンソース・データベースMySQLに格納し、PHPで簡単なプログラミングを行うことで、ウエブ上でオオヒメグモの配列データを自由に検索できるシステムPt_spiderBASEを構築し、公開した(図4; http://www.spider-brh.jp/pt_spiderbase/)。今後も、研究の進展に合わせて、このデータベースの内容を充実させていきたい。
おわりに
本年度は、それぞれのテーマで有効な方法を模索し、試行錯誤しつつ、遺伝子発現の定量解析やバイオインフォマティクス、数理モデリングを行ったが、これらの研究が最終的に向かう先の一つの共通のゴールは、発生可能で、進化可能なバーチャル多細胞集合体の構築である。オオヒメグモのゲノム配列が得られ、多くの研究者がこのクモを使い始める中、オオヒメグモが大きな多様性の単なるひとつとしての価値しか持たないのであれば、このクモを使った研究の発展は望めないであろう。クモの中にどのような価値を見つけられるかが、今問われており、私たちはその価値を、多細胞集合体の発生と進化を支配する理論基盤を理解するためのひとつの入り口として捉えている。一部のクモがはっきりと示す胚発生における調整能力は、バーチャル多細胞集合体のシミュレーションで試したい多細胞体の細胞が持つ性質である。
