年度別活動報告
年度別活動報告書:2012年度
アゲハチョウの食草選択と進化
尾崎 克久(研究員) 龍田 勝輔(奨励研究員)
廣嵜 由利恵(研究補助員)
はじめに
動物は、外界に存在する化合物を味覚・嗅覚として認識している。陸棲生物においては、水溶性・脂溶性の不揮発性化合物を感覚器官が接触して認識するものを味覚、揮発性化合物を非接触的に認識するものを嗅覚と呼ぶ。化学感覚(味覚・嗅覚)は、生物間のコミュニケーションにとって不可欠な機能であり、寄主選択・配偶行動・集団生活の維持・社会性の構築など、生存上の様々な役割を担っている。寄主選択に関わる化学受容の仕組みに変化が生じた場合、それまでとは異なる空間を生息の場として利用する集団が現れ棲み分けが起こり、棲み分けが何世代にもわたって繰り返されることによって種分化へとつながると考えられている。このような変化は、進化の歴史を物語る証拠としてゲノムに刻まれる。動物と環境との関わりにおいて中心的な機能である味覚や嗅覚といった化学受容に関わる分子機構の解明は、多様化・種分化・環境への適応の仕組みを解明するために、最も有力な手がかりになると考えられる。
アゲハチョウの仲間は、他の多くの植食性昆虫と同様に、特定の植物のみを餌として利用する単食性に近い寄主選択をしており、寄主選択とアゲハチョウ科の進化には相関関係が認められる1。卵から孵化したばかりのアゲハチョウの幼虫は、体が小さく移動能力が低いため、広い環境中を歩き回って自力で餌を探索することは困難である。そのため、メス成虫による正確な植物種の識別と産卵場所の選択は、次世代の生存に決定的な影響を与えることになる。メス成虫は産卵の直前に前脚で植物に触れることで、前脚ふ節にある化学感覚子を通じて含有する化合物を感じ取り、その組み合わせによって産卵行動が引き起こされる。前肢ふ節の感覚子数は雌雄間で大きな差があり、圧倒的にメスに多く、メスのみが行う行動、つまり産卵において重要な役割を持つことが知られている。ナミアゲハ(Papilio xuthus)では、主な食草であるウンシュウミカンの葉から産卵刺激物質として10種類の化合物が単離されている2。他にも数種のアゲハ類で産卵刺激物質が明らかにされており、これら化合物の構造についてアゲハ種間で比較すると、類似性が認められる。このことから、植物の系統的近縁性とは無関係に、植物に含まれている化合物の類似性が寄主転換の可能性を支え、食性の進化を可能にしたのではないかと考えられている3。これまでに報告されている産卵刺激物質は全て不揮発性で、前脚ふ節で接触して感じていることから、アゲハチョウは前脚で「味」として認識していると考えられている。前脚での味の感じ方に変化が生じた場合、それまでとは異なる植物を選択する集団が現れて、住み分けによる隔離を出発点とする同所的種分化という現象を引き起こしたと考えられる。
ミカン科食性のアゲハチョウ間であっても、種ごとに産卵刺激物質として認識する化合物の組み合わせが異なるため、種ごとに特徴的な多様な味覚受容体を持っていることが予測される。また、味覚受容体が化合物を認識するためには、化合物が化学感覚子内のリンパ液を通り抜けて受容体に到達する必要があるため、化合物を結合して運搬する役割を持つタンパクが必要になると考えられる。味覚受容体と化合物結合タンパクを中心とする産卵刺激物質受容システムに関わる遺伝子群を解明し、複数種間で比較することができれば、食草転換を原動力として起きた進化という現象のメカニズムを理解する重要な手がかりになると考えた。
昆虫の味覚に関する研究が本格的に始まったのは30年近く前に遡るが4、味覚受容体としては2000年になって初めて7回膜貫通型受容体(以下7TMRと略)が報告された5。化学受容の7TMRは一次構造の多様性が高く、脊椎動物から報告されている味覚・嗅覚の7TMRに対する類似性を手がかりとした探索は困難であり、ショウジョウバエの全ゲノム配列の情報科学的解析によって候補遺伝子ファミリーが同定された。これまでに7種の昆虫の全ゲノムが解読されているが、味覚受容体遺伝子はゲノムからコンピュータで解析されたものに限られている。興味深いことに、これまでに報告されている昆虫化学感覚受容体は、昆虫間でも類似性に乏しく、それぞれの生活史に合わせて独自の多様化をしている可能性が示唆される。昆虫の味覚受容体は極端に発現量が少ないため解析は困難を極め、機能が解明されているものはまだ少ない6,7。化合物結合タンパクについて、味覚器官で発現するものが見つかっており8、食草の選択に重要な役割を持つものも報告されている9。ナミアゲハでは、Chemosensory protein (CSP) がゲノムの特定の領域にクラスターしており10、カイコとシンテニーがある事を確認している。また、多種昆虫から化合物結合タンパクの遺伝子が報告され、情報が蓄積しつつある。
本研究は、主たる食草の産卵刺激物質が明らかにされているアゲハチョウ科昆虫を材料として用い、メス成虫前脚ふ節に発現する味覚受容体遺伝子及び化合物結合タンパク遺伝子をクローニングし、その機能と特徴の解明を目的として取り組んでいる。
昨年度までの取り組みで、ナミアゲハからメス成虫前脚ふ節に発現の特異性を示す7回膜貫通型タンパク質遺伝子(PxutGr1)を発見し、バキュロウイルス発現系を用いたカルシウムイメージング法により産卵刺激物質の一つであるSynephrine(シネフリン)に特異的に応答する受容体である事を解明している。また、RNAi法を用いた機能阻害と電気整理実験・産卵行動実験に取り組んだ結果、PxutGr1の発現を阻害したナミアゲハでは、Synephrineに対する感度が低下し、産卵行動が抑制されることを確認している。これらの結果から、ゲノムが読まれていない昆虫から初めて味覚受容体遺伝子を同定し、産卵という本能行動に関与していることを明らかにした11。また、電気生理実験により、産卵行動を引き起こすために複数の産卵刺激物質を同時に認識する必要がある仕組みについて、神経プロファイルを解明している12。
今年度は、ナミアゲハと同様に産卵刺激物質が解明されているクロアゲハとシロオビアゲハについて、産卵行動を引き起こす神経プロファイルの解明を試みた。また、ナミアゲハとシロオビアゲハのドラフトゲノムを用い、新規の味覚受容体遺伝子を発見した。
結果と考察
1. アゲハチョウの産卵刺激神経プロファイル
アゲハチョウ科昆虫は狭食性を示し、種によってミカン科、セリ科、クスノキ科、ウマノスズクサ科の植物を寄主植物としている。その厳密な寄主選択は雌成虫の産卵行動時に行われており、雌成虫は前肢ふ節に存在する感覚子で寄主植物葉に含まれる化合物を認識した後、葉に産卵する。アゲハチョウ科の寄主選択メカニズムの解明は、これまで化学生態学分野の研究を中心として、寄主植物に含まれる化合物(産卵刺激物質)を同定することで行われてきた。その中でも、ナミアゲハ、シロオビアゲハ、クロアゲハの3種は複数の産卵刺激物質が同定されている(それぞれ、10、5、6種類)2,13,14。これら3種のチョウの産卵刺激物質に共通しているのは、それらが単独では産卵活性を示さず、複数の産卵刺激物質が混合して初めて産卵活性を持つことである。しかしながら、複数の産卵刺激物質がふ節感覚子でどのように受容され、産卵行動を誘起するのか、そのメカニズムは未解明であった。
本研究では、ナミアゲハのふ節感覚子の産卵刺激物質受容メカニズムの解明を行ってきた。産卵刺激物質に対する電気生理実験、産卵行動実験を行った結果、ナミアゲハふ節感覚子内の3種類の味細胞が産卵刺激物質に特異的に応答し、それらの味細胞が同時に興奮(神経発火)することが産卵行動誘発に必要であることがわかった12。
本年度は、ナミアゲハに加え、産卵刺激物質が明らかとなっているクロアゲハ、シロオビアゲハの電気生理学解析を行い、2種の産卵刺激物質受容メカニズムの解明を目指した。
2. 電気生理実験による発現プロファイルの比較
クロアゲハ前肢ふ節感覚子のミカン葉抽出物に対する電気生理応答を調べた結果、ミカン葉抽出物に対して、その高さの違いにより3種類のスパイク(大、中、小)が発生した(図1)。異なる高さのスパイクは異なる味細胞の応答によって発生するため、3種類のスパイクが発生することは、同時に3種類の味受容細胞が応答していることを示す。よって、クロアゲハは最低でも3種類の味細胞がミカン葉抽出物に含まれる化合物を受容すると考えられる。
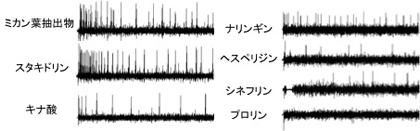
図1 電気生理応答例(クロアゲハ雌)
クロアゲハの産卵刺激物質としてウンシュウミカン葉から同定されている6 種類の化合物に対する電気生理学的応答を調べた結果、プロリン以外の5種類の化合物で応答を確認できた(図2)。スタキドリンやキナ酸は高いスパイクと低いスパイクを同時に発生させており、これらの化合物は2種類の味細胞によって受容されていることがわかった。また、ナリンギン、ヘスペリジン、シネフリンは、1種類のスパイクを発生させ、そのスパイクは、スタキドリン、キナ酸によって発生する2種類のスパイクに対し、中間の高さであった。これらの結果より、クロアゲハでは5種類の産卵刺激物質に対し3種類の味細胞が応答している可能性が示唆された。さらに、発生するスパイクの頻度(記録開始から2秒間のスパイク数)は、化合物の濃度の上昇に伴い高くなるため、これらの化合物に対する味細胞の応答によって発生するスパイクであることが明らかとなった(図2)。
シロオビアゲハは5種類の産卵刺激物質がウンシュウミカン葉から同定されている13。これら5種類の化合物の電気生理応答を調べた結果、すべて化合物で応答が確認できたが(図3)、プロリンおよびコリンにおいて応答が確認できたのは、わずか数例のみであった。また、ミカン葉抽出物に対してはナミアゲハ・クロアゲハと同様に3種類のスパイクを発生させ、かつスタキドリンなどは単一の化合物であるものの2種類のスパイクを発生させることがわかった(データ未掲載)。しかし、シロオビアゲハの電気生理実験結果は、データ数が少ないためスパイク解析を行うにはデータ数が少なく、不十分であるため、今後さらなる実験が必要である。
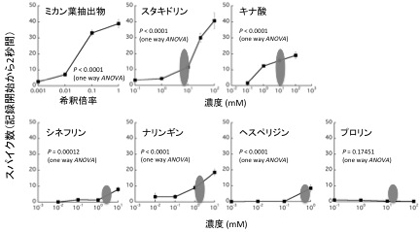
図2 ミカン葉抽出物、産卵刺激物質に対するスパイク頻度(クロアゲハ)
灰色円は各化合物のミカン葉に含まれる推定濃度を示す14
スパイク数は記録開始から2秒間に発生したすべてのスパイクの総数を示す
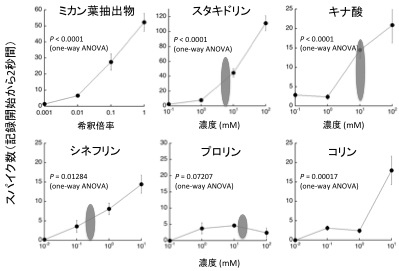
図3 ミカン葉抽出物、産卵刺激物質に対するスパイク頻度(シロオビアゲハ)
灰色円は各化合物のミカン葉に含まれる推定濃度を示す14
スパイク数は記録開始から2秒間に発生したすべてのスパイクの総数を示す
以上の結果より、クロアゲハ、シロオビアゲハがナミアゲハと同様に3種類の味細胞によって産卵刺激物質を認識していることが予想される。しかしながら、クロアゲハとシロオビアゲハの電気生理学解析はデータ数が少ないため、今後さらなる解析が必要である。さらには産卵行動実験を行い、産卵刺激物質を受容する細胞の応答パターンと行動結果を比較する必要があるため、来年度はこれらの研究に着手したい。
また、これら3種のアゲハチョウ以外に、ミカン科を食草するナガサキアゲハや、食草が異なるキアゲハ(セリ科植物)、ジャコウアゲハ(ウマノスズクサ科植物)などの電気生理学的解析、産卵行動実験を行うことにより、アゲハチョウ科昆虫における寄主転換による種分化の可能性を考察できればおもしろいと考えている。
3. アゲハチョウのドラフトゲノムと次世代型シーケンサーを用いた遺伝子探索
文部科学省科学研究費新学術領域研究「ゲノム支援」の協力を得て、ナミアゲハとシロオビアゲハのゲノム解読を行った。ドラフトゲノムから新規の味覚受容体遺伝子を検出し、比較解析を行った。この取り組みにより、チョウの仲間の間で保存されていると考えられる味覚受容体遺伝子を3種類発見した。今年度導入された次世代型シーケンサー MiSeq を用い、RNA-seq による発現の確認を行った。ドラフトゲノムと MiSeq のデータから、未発見であった化合物結合タンパクの遺伝子を多数検出した。
4. アゲハチョウのドラフトゲノム
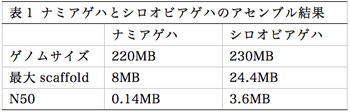
ゲノム支援により、2011年度にナミアゲハ、2012年度にシロオビアゲハのゲノム塩基配列を次世代型シーケンサーで読み、アセンブルを行った。ナミアゲハは Roche 454 GS FLX で読んだシングルエンド(一方向から塩基配列を読んだもの)のデータを Newbler (Roche社のシーケンサーに標準添付されているアセンブルソフト)でアセンブルし、シロオビアゲハは illumina HiSeq 2000 で読んだペアエンド(両方向から向かい合わせに塩基配列を読んだもの)のデータを platanus (東工大伊藤武彦教授が開発中のアセンブルソフト)でアセンブルした。データ量はどちらも、全ゲノムの200倍を超える重複率である。
ナミアゲハのゲノムは、約220MBの大きさであると推定された。scaffold (結合したゲノムの配列を、部分的なギャップを残しつつ連結させたもの)は最大で8MB(メガベース: 百万塩基)、N50 (結合された scaffold を大きい方から順に並べていって、その合計サイズが全ゲノムの半分を超えたときの scaffold の大きさ)の値は0.14MBであった(表1)。ゲノム配列のアセンブル結果は、N50の値を精度の判断基準として用いるが、一般的には1MBを超えることが望ましいとされる。
シロオビアゲハのゲノムは、約230MBの大きさであると推定された。最大の scaffold は約24.4MB、N50の値は約3.6MBと、大変良好な結果が得られた(表1)。platanus は、アセンブルで分岐が起きる際に、k-mer の値(アセンブルの計算をする際に、塩基配列を一時的に小さな単位に分解する長さ)を可変させることで分岐を乗り越えることができるため、ヘテロ性が高いゲノムのアセンブルに強いという特徴があり、この機能が好影響したのではないかと考えられる。
5. 新規味覚受容体遺伝子の探索
新規の遺伝子を探索する際、PSI-BLAST 15を使って検出したい遺伝子ファミリーのアミノ酸コンセンサス配列を作成し、そこから算出した位置スコア行列を用いて tBLASTn でのゲノム配列の検索を行い、遺伝子の断片を探索する手法が一般的である。昨年度はこの方法でナミアゲハのゲノム配列から遺伝子探索に取り組んだ。しかし、多様性が高い昆虫化学受容体の場合、検索でヒットしない虞れがあるため、今年度は別の方法を用いた。
まず、EMBOSS 16という解析パッケージに含まれる getorf というツールを用い、ゲノム配列から10個以上連続してアミノ酸配列に置き換えることができる部分を網羅的に検出した。その結果、ナミアゲハのゲノムからは、約14万5千個、シロオビアゲハのゲノムからは約17万個のアミノ酸配列が得られた。これらの配列を用い、昆虫味覚受容体遺伝子に対して BLASTP 17検索を行い、ヒットした query に絞り込んだ。絞り込んだアミノ酸配列で nr (全アミノ酸初列情報のデータベース)に BLASTP 検索を行い、候補遺伝子を探索した。既知遺伝子のデータベースだけでは検出できないくらいホモロジーが低い味覚受容体遺伝子が存在する可能性があるので、ナミアゲハとシロオビアゲハ間で新規の味覚受容体候補遺伝子を使って相互にゲノム配列から遺伝子領域を検索した。
この方法で、ナミアゲハのゲノムからは既知の PxutGR1 を含む37個、シロオビアゲハのゲノムからは51個の味覚受容体候補遺伝子が見つかった(表2)。ナミアゲハのゲノム配列は、contig (ギャップが無い状態に結合した塩基配列の塊)や scaffold (ギャップを残しつつ contig を連結させたもの)のサイズが小さく、多くのギャップが残っているため、検出できていない遺伝子も残されているのではないかと考えられる。
同じ方法で検出したCSPの数を表1に示す。OBPについては、両種とも約40個の候補配列が見つかったが、データベースとのホモロジーが低く判断が難しいものが多いため、今後詳細な解析が必要である。
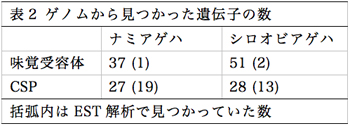
これまでに行ってきた EST 解析では検出できなかった遺伝子が、ドラフトゲノムから多数発見できたことから、ドラフトゲノムは新規の遺伝子探索に有益な手段であると判断できる。特に昆虫の味覚受容体遺伝子は、発現量が低く一次配列の類似性も低い場合が多いため、実験的手法で検出することが困難であることから、ゲノム情報の充実によりこの分野の研究が飛躍的に推進される可能性に期待がかかる。
発見した味覚受容体候補遺伝子をゲノム配列にマッピングすると、単独の scaffold に複数の遺伝子が配置されるものがあり、小規模なクラスターを形成している領域があることが解る。 ナミアゲハのCSP遺伝子は、約76KBの領域に17個の遺伝子がクラスターしており、カイコゲノムと Synteny (遺伝子の配置パターンなど、ゲノムの構造的な類似性)があることを報告している10。シロオビアゲハのCSP遺伝子も同様のクラスターを形成しており、ナミアゲハと高いホモロジーがあることが観察された(データ未掲載)。ナミアゲハ・シロオビアゲハ・クロアゲハの3種間で、CSP遺伝子の同義置換と非同義置換の割合を比較すると、進化速度に遺伝子間で大きなばらつきがあり、高度に保存されているものや積極的に変化しているものがあることが解った(データ未掲載)。これらCSP遺伝子領域のゲノム構造の詳細については、別の機会に報告する予定である。
6. RNA-seq による発現の確認
2012年9月に次世代型シーケンサー illumina MiSeq が導入されたので、ナミアゲハとシロオビアゲハのメス成虫ふ節の RNA-seq を行った。RNA-seq とは、次世代型シーケンサーの高出力性能を活かし、どの様な生物でも遺伝子発現産物の網羅的な解析が可能になる実験手法である。BRH への MiSeq 導入時は、ソフトウエアのみ Ver. 2 でハードウエア本体は Ver. 1 という変則的な状態であったため、リード長は250塩基まで読めるものの生成されるクラスターの数は最大で700万個というスループットであった(2013年1月18日にハードウェアバージョンアップが完了)。
illumine 社の TrueSeq RNA Preparation kit を用い、メーカーのプロトコールに従ってシーケンスライブラリーを作成し、MiSeq で塩基配列を決定した結果、両種のライブラリーで約500万個のクラスターが形成され、クラスターを両方向から読んだので約1,000万リード x 250塩基のデータが得られた。
得られたデータを Trinity 18という発現産物の配列データ結合用ソフトを用いてアセンブルした結果、ナミアゲハからは約60万個、シロオビアゲハからは約45万個の contig (結合した塩基配列の塊)が得られた。平均の contig 長はどちらも約600塩基という短さであった。これらの結果から、完全長のmRNAが再現されていないものが大部分であると予想される。Trinity で得られた contig を用い、PCAP というゲノムアセンブラーで第二段階の結合を行った結果、それぞれ約17万個と13万個に contig 数が減少し、平均長は約800塩基に改善された。
これらアセンブル結果の contig をデータベース化し、新規に見つかった味覚受容体候補遺伝子の配列を用いて tBLASTn 検索を行った結果、両種ともに12個の候補配列がヒットしたことから、こられに関しては少なくともメス成虫の前脚ふ節で発現していると考えられる。しかし、どの contig も断片的であり、味覚受容体遺伝子の全長配列が得られるものはなかった。今後は RNA-seq データをアセンブルする前に、塩基配列データのクオリティ値が低い部分を削除する、短すぎるリードを除去するなど、アセンブルに用いる塩基配列データの精度を高める工夫を加える必要があると思われる。これまでにEST解析で部分配列が見つかった味覚受容体遺伝子(7回膜貫通型のタンパク質)は、通常の実験方法では5'末端側の配列決定に苦労した経験があるので、RNA-seq で完全長の発現産物データが得られるようになれば、新規遺伝子探索と全長配列決定の速度が飛躍的に向上することに期待できる。
7. 保存性の高い味覚受容体遺伝子の発見
昆虫の味覚受容体遺伝子は多様性が高く、現在までに知られているものに関しては種を超えて類似性の高いものはごく希である。また、嗅覚受容体の Orco 19(オルコ = 全ての嗅覚受容体と共発現する、全昆虫で高度に保存されている遺伝子: 旧名称は Or83b ファミリー)の様に多くの昆虫種間で保存されているものも見つかっていない。
本研究で見つかった新規の味覚受容体候補遺伝子を2種間で比較すると、3つがナミアゲハとシロオビアゲハの間でアミノ酸配列が90%を超える高いホモロジーを示した。これらを用いて nr データベースに対して BLASTP 検索を行うと、オオカバマダラ(Danaus plexippus)の味覚受容体と80%を超えるホモロジーを示した(図4)。ショウジョウバエやハマダラカといった、目レベルで異なる昆虫の遺伝子にも、約60%の高いホモロジーがあった(データ未掲載)。
この様に高度に保存されている遺伝子は、少なくともチョウの間で共通する重要な役割を持っている可能性がある。今後、発現部位等を調査し、食草認識との関わりが示唆される場合には機能解析に取り組みたい。

図4 高保存性味覚受容体のアライメント
Pxut: ナミアゲハ、Ppol: シロオビアゲハ、Dple: オオカバマダラ、Gr: 味覚受容体
この他、Gr22とGr24が、同様に3種間で高い類似性を示す
おわりに
今年度の取り組みにより、クロアゲハやシロオビアゲハもナミアゲハと同様に3種類の味覚感覚神経が同時に発火することが、産卵行動の誘発に必要であることが示された。「産卵誘導の神経プロファイル」という仕組みが種を超えて共通しているということは、食草の情報の入り口にあたる味覚受容体に変化が生じた場合に、応答する化合物が変化することになるが、脳へ伝わる産卵誘導刺激は全く同じものであるという可能性が考えられる。最小限の遺伝子変異で食性が変わるという可能性を示唆する、興味深い発見であると言える。
ゲノムのアセンブル結果は、ナミアゲハとシロオビアゲハの2種間で精度に大きな差があったが、どちらも新規の遺伝子探索には十分に役立ったことから、荒いドラフトゲノムであっても研究のツールとして有益であると言える。ナミアゲハとシロオビアゲハで比較解析が行えるようになったことで、保存性の高い味覚受容体を発見できたことは興味深い。多くのアゲハチョウの仲間のゲノムを解読し、文房具のように自在に利用できるようになれば、この分野の研究が飛躍的に推進され、昆虫と植物の間にある相互作用を総合的に理解できることに期待できる。MiSeq のような小型シーケンサーでも、アゲハチョウのようにゲノムサイズが比較的小さい昆虫は、単独の研究室でゲノムを読むことが可能である。ドラフトゲノムをデータベース化し、情報を効率よく利用できる仕組みを構築することが、今後の取り組みとして重要になるであろう。
TALEN に代表されるゲノム編集技術の発展は目覚ましく、いわゆる"モデル生物"と"非モデル生物"の差はかつて無いほどに小さなものになっている。この様な新しい技術を取り入れ、これまでに確立している電気生理実験や行動解析と組み合わせ、効率的に機能解析が行えるように取り組んでいきたいと考えている。
謝辞
本研究の遂行にあたり、吉川寛非常勤顧問(BRH)、谷村禎一准教授(九大)、Frederic Marion-Poll教授(INRA)に多大な協力と助言を頂いた。
引用文献
- 1 Thompson, J. N. Evolutionary genetics of oviposition preference in swallowtail butterflies. Evolution 42, 1223-1234 (1988).
- 2 Ohsugi, T., Nishida, R. & Fukami, H. Multi-component system of oviposition stimulants for a Rutaceae-feeding swallowtail butterfly, Papilio xuthus. Applied Entomology and Zoology 26, 29-40 (1991).
- 3 Feeny, P. in Swallowtail Butterflies: Their Ecology and Evolutionary Biology (eds JM Scriber, Y. Tsubaki, & RC. Lederhouse) 9-15 (Scientific Publishers, 1995).
- 4 Tanimura, T., Isono, K., Takamura, T. & Shimada, I. Genetic dimorphism in the taste sensitivity to trehalose in Drosophila melanogaster. J. Comp. Physiol. 141, 433-437 (1982).
- 5 Clyne, P. J., Warr, C. G. & Carlson, J. R. Candidate taste receptors in Drosophila. Science 287, 1830-1834, doi:8337 [pii] (2000).
- 6 Jones, W. D., Cayirlioglu, P., Kadow, I. G. & Vosshall, L. B. Two chemosensory receptors together mediate carbon dioxide detection in Drosophila. Nature 445, 86-90 (2007).
- 7 Chyb, S., Dahanukar, A., Wickens, A. & Carlson, J. R. Drosophila Gr5a encodes a taste receptor tuned to trehalose. Proc Natl Acad Sci U S A 100 Suppl 2, 14526-14530 (2003).
- 8 Galindo, K. & Smith, D. P. A large family of divergent Drosophila odorant-binding proteins expressed in gustatory and olfactory sensilla. Genetics 159, 1059-1072 (2001).
- 9 Matsuo, T., Sugaya, S., Yasukawa, J., Aigaki, T. & Fuyama, Y. Odorant-binding proteins OBP57d and OBP57e affect taste perception and host-plant preference in Drosophila sechellia. PLoS Biol 5, e118, doi:06-PLBI-RA-0167R3 [pii] 10.1371/journal.pbio.0050118 (2007).
- 10 Ozaki, K., Utoguchi, A., Yamada, A. & Yoshikawa, H. Identification and genomic structure of chemosensory proteins (CSP) and odorant binding proteins (OBP) genes expressed in foreleg tarsi of the swallowtail butterfly Papilio xuthus. Insect Biochem Mol Biol 38, 969-976, doi:S0965-1748(08)00133-1 [pii] 10.1016/j.ibmb.2008.07.010 (2008).
- 11 Ozaki, K. et al. A gustatory receptor involved in host-plant recognition for oviposition of the butterfly, Papilio xuthus. Nature communications 2:542 (2011).
- 12 Ryuda, M. et al. Gustatory sensing mechanism coding for multiple oviposition stimulants in the swallowtail butterfly, papilio xuthus. J Neurosci 33, 914-924, doi:10.1523/JNEUROSCI.1405-12.2013 33/3/914 [pii] (2013).
- 13 Murakami, T. Studies on the relationships between host-plant acceptability and plant constituents in host selection by a swallowtail butterfly, Papilio polytes. Grad Sch Biosp Sci 42, 58-59 (2003).
- 14 Honda, K. Identification of host-plant chemicals stimulating oviposition by swallowtail butterfly, Papilio protenor. Journal of Chemical Ecology 16, 1127-1135 (1990).
- 15 Altschul, S. F. et al. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acids Res 25, 3389-3402, doi:gka562 [pii] (1997).
- 16 Rice, P., Longden, I. & Bleasby, A. EMBOSS: the European Molecular Biology Open Software Suite. Trends Genet 16, 276-277, doi:S0168-9525(00)02024-2 [pii] (2000).
- 17 Mount, D. W. Using the Basic Local Alignment Search Tool (BLAST). CSH Protoc2007, pdb top17 (2007).
- 18 Grabherr, M. G. et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nat Biotechnol 29, 644-652, doi:10.1038/nbt.1883 nbt.1883 [pii] (2011).
- 19 Vosshall, L. B. & Hansson, B. S. A unified nomenclature system for the insect olfactory coreceptor. Chem Senses 36, 497-498, doi:bjr022 [pii] 10.1093/chemse/bjr022 (2011).
