年度別活動報告
年度別活動報告書:2014年度
分子系統から生物進化を探る 3-2. 六脚類の起源と節足動物の系統進化過程の解明
蘇 智慧(主任研究員) 和智仲是(奨励研究員)
佐々木綾子(研究補助員) 宮澤秀幸(大阪大学大学院生 招聘研究員)
南紘彰(大阪大学大学院生)
はじめに
記載種数において動物界最大の分類群である現生の節足動物門は、鋏角亜門 (Chelicerata; 以下、鋏角類)・多足亜門 (Myriapoda; 以下、多足類)・甲殻亜門 (Crustacea; 以下、甲殻類)と六脚亜門 (Hexapoda; 以下、六脚類) の4亜門に分類される。この節足動物門に属する生物は、地球上の至る所に分布しており人類にとっても身近なものが多い。しかし節足動物門内の系統関係について多くの点が未だ明確になっていない。多足類の系統的位置については多足類と鋏脚類が姉妹群を形成するMyriochelata説と多足類が汎甲殻類(甲殻類+六脚類)と姉妹群を作るMandibulata(大顎類)説が提唱されている。近年の分子系統学的研究では大顎類説がより支持されている。しかしその多くが信頼性の低い樹形から得られた結論のため、検討の余地が残されている。六脚類と甲殻類の関係についてはミトコンドリア遺伝子による研究では無翅昆虫の内顎類(カマアシムシ目+トビムシ目+コムシ目)が甲殻類の一部より前に分岐した可能性が示唆され、六脚類の単系統性も疑問視されていた。しかし、最近の多くの研究結果はその可能性を否定している8,9)。
各亜門における「綱」間・「目」間の系統関係についても明確になっていないところが多く残されている。我々はこれまでに複数の核タンパク遺伝子を用いて、六脚類と多足類の目間の系統関係の解明を行ってきた8,10)。六脚類については、これまで提唱されていた内顎類・無尾類・無眼類の単系統性を否定し、分子系統学的にカマアシムシ目が六脚類において最も先に分岐した系統であることを示した。また有翅昆虫の系統関係についても、多新翅類の単一起源を強く支持する系統仮説を分子情報に基づいて初めて提唱し、完全変態類昆虫の目間の系統関係をほぼ完全に明らかにした。多足類については、ムカデ綱 Chilopoda・ヤスデ綱 Diplopoda・コムカデ綱 Symphyla・エダヒゲムシ綱 Pauropodaのうち、コムカデ綱が最も祖先的な系統であることを明らかにした。さらに、その分岐はこれまで考えたより遙かに古く、カンブリア紀初期に遡ることが判明した。また、変態様式の祖先状態の推定を行った結果、多足類の祖先種は1)半増節変態を行っていたこと、2)体節と脚の数が少なかったことが示唆された11)(図3)。しかし、これまでの研究によって六脚類と多足類の系統関係に対する理解が大きく前進したとはいえ、未解明な部分が多く残されている。多くの研究が行われて来たにも関わらず、節足動物門の系統関係についての十分な理解が進まない主な原因の一つは解析に使用される分子情報の不適切性と量の不足であると考えられる。そこで、我々は昨年度から次世代シークエンサーを活用した大量の核タンパク遺伝子情報を用いたPhylogenomics(ゲノム系統学的解析)に取り組んでいる。このような背景のもと、本年度行った多足類についての研究結果について報告する。
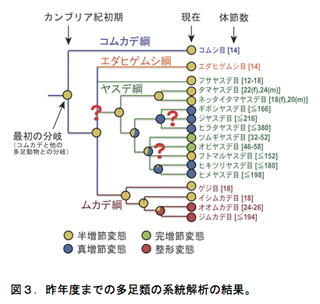
結果と考察
1)解析サンプルの選択
多足類は、ムカデ綱(5目)・ヤスデ綱(16目)・コムカデ綱(1目)・エダヒゲムシ綱 (2目)の4綱からなる。これまで多足類の綱レベルの系統関係についてはさまざまな仮説が提唱されてきた。核タンパク質遺伝子RNA polymerase II largest subunit (RPB1), RNA polymerase II second largest subunit (RPB2) とDNA polymerase δ catalytic subunit (DPD1)を用いて系統関係を推定したところ、コムカデ綱が最初に分岐し残り3綱(ムカデ綱・ヤスデ綱・エダヒゲムシ綱)が一つの系統群であることが示唆された(図3)。コムカデ綱とその他の3綱が姉妹群になる点で、我々の結果はこれまでに提唱されてきた系統仮説と異なっている。その一方で、ムカデ綱とヤスデ綱の目レベルの系統関係では形態に基づく系統仮説と矛盾しない結果になった。しかし、エダヒゲムシ綱・ヤスデ綱・ムカデ綱の綱レベルの関係やヤスデ綱の目レベルの関係の一部については検討の余地が残されている。これらの系統関係を詳細に明らかにすることは、節足動物門において多足類特有の変態様式の起源や進化過程についての理解につながる。多足類の目レベルの系統関係を明らかにするために、それぞれの系統群から代表種(表4)を選定した。
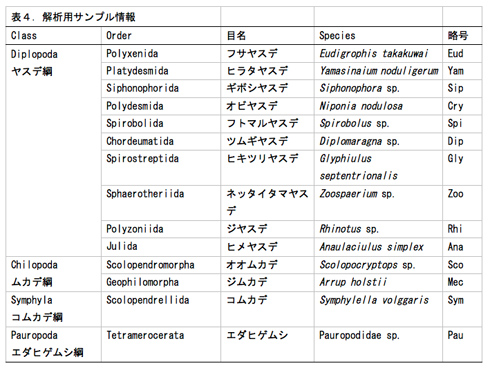
2)次世代シークエンサーによる配列情報の取得と解析
必要な材料(計14種:ヤスデ10種、ムカデ2種、コムカデとエダヒゲムシはそれぞれ1種; 表4)について、昨年度までにcDNAを得ている。それらのcDNAを元に次世代シークエンサー用ライブラリーを作成した(Nextera)。得られたライブラリーを次世代シークエンサーで解析した結果、約400万〜1800万の配列が得られた(表5, 図4)。これらの配列をもとにアセンブル(CLC Genomics Workbench)した結果、材料によって得られたコンティグ数にばらつきが見られた(図4)。
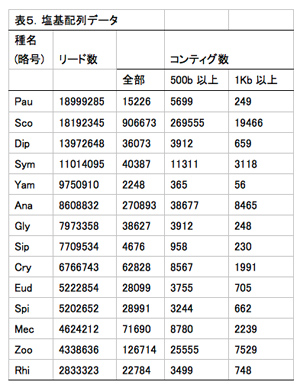
解析に用いた配列数が少ないためにコンティグ数も少なくなっている可能性があるが、リード数とコンティグ数に必ずしも比例していなかった。例えば、オオムカデ(Sco)とエダヒゲムシ(Pau)は、ともに1800万以上のリードが得られたが、コンティグの数は大きく異なっている(図4)。これらの結果は、解析に用いるcDNAの質が最終的に得られる配列情報の質に大きく影響を与えることを示唆している。今後、目標とするコンティグ数(種につき数万)と質(1000塩基以上)を得るために、明らかに情報が足りない種については新たなサンプルを採集してRNA抽出の段階からやり直すことを検討している。
3)相同遺伝子(オーソログ遺伝子)配列の抽出
得られた遺伝子の配列は必ずしもそのまま全て系統解析に使用できるとは限らない。遺伝子配列の中には系統解析に不適切なものが多く含まれているからである。大量の遺伝子配列から、解析に適したそれぞれの種で相同な遺伝子配列を得るために、それ以外の配列をとり除く必要がある。まず、それぞれのコンティグ配列をアミノ酸配列に翻訳する。複数の節足動物(全ゲノム配列が分かっているもの)に共通して存在する単一コピーの相同遺伝子を参考配列として、類似性の高いコンティグ配列を検索し、系統解析に使用するための候補配列を得る(HaMStR)。
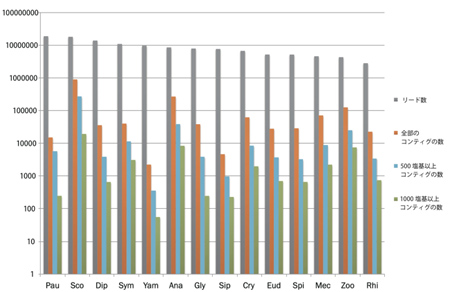
図4.各種から得られた塩基配列のリード数とアセンブルした後のコンティグの数。横軸にある種名の略号は表4と表5のものと一致する。
各種から抽出した候補配列を遺伝子座ごとにアライメントを行う(MAFFT)。その後、系統推定の際にノイズとなるような配列を取り除いた(ALISCORE, MARE)。得られた配列情報から予備的に相同遺伝子配列を抽出した(図5)。リード数、コンティグ数が十分でも、基準に合う配列が少なすぎる場合が多く見られた。今後、取捨選択の閾値を変えるなどの工夫が必要である。
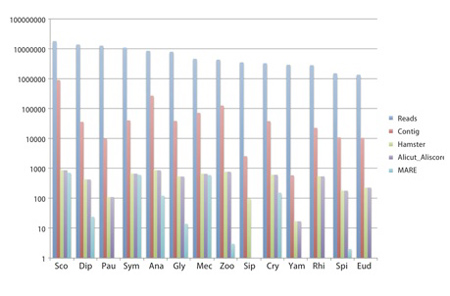
図5.系統解析に適した遺伝子配列の抽出。横軸にある種名の略号は表4と表5のものと一致する。
本年度の研究によって、単純に配列情報を増やすだけでなく、より適した抽出方法を確立する必要があることが明らかになった。最終的には、およそ500遺伝子座でゲノム系統学的解析を行うことを目標にしている。また、配列の持つ系統情報が遺伝子の機能によって偏るかどうか、系統関係を推定する際に適した情報を持つ遺伝子に共通の機能はあるか、といったことを検討できたらと考えている。
おわりに
これまでに複数の核タンパク遺伝子座の情報を用いて、昆虫類や多足類を中心に節足動物門の系統進化の解明を行い、その理解に大きく貢献してきた。しかし、現在でも未解明の問題は少数の遺伝子では解明することが困難であることが予想され、網羅的な遺伝情報を用いたゲノム系統学的解析が必要であると考えられる。近年、様々な分類群でそのようなゲノム系統学的解析が盛んに行われており、昆虫類の結果もごく最近発表された12)。多足類のゲノム系統学的解析を完成させるために、今後、新たな試料の採集•配列情報の新規獲得を行っていきたい。多足類の綱レベルと目レベルの系統関係を明らかにすることで、多足類の変態様式と体節増加の進化過程を解明したい。最終的には、さらに鋏角類や汎甲殻類の情報も加えて節足動物門全体の系統解析を行い、その進化を探りたい。
引用文献
- 1) Herre E.A., Jander K.C. and Machado C.A. (2008) Evolutionary ecology of figs and their associates: recent progress and outstanding puzzles. Ann. Rev. Ecol. Evol. Syst. 39: 439–458.
- 2) Su Z.-H., Iino H., Nakamura K. Serrato A. and Oyama K. (2008) Breakdown of the one-to-one rule in Mexican fig-wasp associations inferred by molecular phylogenetic analysis. Symbiosis 45: 73–81.
- 3) Cornille, A., Underhill, J.G., Gruaud, A., Hossaert-McKey, M., Johnson, S.D., Tolley, K.A., Kjellberg, F., von Noort, S. and Proffit, M. (2012) Floral volatiles, pollinator sharing and diversification in the fig-wasp mutualism: insights from Ficus natalensis, and its two wasp pollinators (South Africa). Proc. R. Soc. B 279: 1731–1739.
- 4) Cruaud, A. et al. (2012) An extreme case of plant–insect codiversification: figs and fig-Pollinating wasps. Syst. Biol. 61: 1029–1047.
- 5) Azuma H., Harrison R.D., Nakamura K. and Su Z.-H. (2010) Molecular phylogenies of figs and fig-pollinating wasps in the Ryukyu and Bonin (Ogasawara) islands, Japan. Genes Genet. Syst. 85: 177–192.
- 6) Kusumi, J., Azuma, H., Tzeng, H.-Y., Chou, L.-S., Peng, Y.-Q., Nakamura, K., and Su, Z.-H. (2012) Phylogenetic analyses suggest a hybrid origin of the figs (Moraceae: Ficus) that are endemic to the Ogasawara (Bonin) Islands, Japan. Mol. Phylogenet. Evol. 63: 168–179.
- 7) Xiao, J.-H. et al. (2013) Obligate mutualism within a host drives the extreme specialization of a fig wasp genome. Genome Biol. 14: R141.
- 8) Sasaki, G., Ishiwata, K., Machida, R., Miyata, T. and Su, Z.-H. (2013) Molecular phylogenetic analyses support the monophyly of Hexapoda and suggest the paraphyly of Entognatha. BMC Evol. Biol. 13:236.
- 9) Regier J.C., Shultz J.W., Zwick A., Hussey A., Ball B., Wetzer R., Martin J.W., Cunningham C.W. (2010) Arthropod relationships revealed by phylogenomic analysis of nuclear protein-coding sequences. Nature 463: 1079–1083.
- 10) Ishiwata K., Sasaki G., Ogawa G., Miyata T. and Su Z.-H. (2011) Phylogenetic relationships among insect orders based on three nuclear protein-coding gene sequences. Mol. Phylogenet. Evol. 58: 169–180.
- 11) Miyazawa, H., Ueda, C., Yahata, K., and Su, Z.-H. (2014) Molecular phylogeny of Myriapoda provides insights into evolutionary patterns of the mode in post-embryonic development. Scientific Reports 4:4127.
- 12) Misof, B. et al. (2014) Phylogenomics resolves the timing and pattern of insect evolution. Science 346: 763-767.
