年度別活動報告
年度別活動報告書:2013年度
分子系統から生物進化を探る 3-2. 六脚類の起源と節足動物の系統進化過程の解明
蘇 智慧(主任研究員) 和智仲是(奨励研究員)
佐々木綾子(研究補助員) 宮澤秀幸(大阪大学大学院生)
はじめに
現生の節足動物門は、記載種数において動物界最大の分類群であり、鋏角亜門 (Chelicerata; 以下、鋏角類)・多足亜門 (Myriapoda; 以下、多足類)・甲殻亜門 (Crustacea; 以下、甲殻類)と六脚亜門 (Hexapoda; 以下、六脚類) の4亜門に分類される。この節足動物門に属する生物は、地球上の至る所に分布しており人類にとっても身近なものが多い。しかし、節足動物門内の系統関係は未だ多くの点が明確になっていない。例えば、従来六脚類は甲殻類よりも多足類と近縁であると考えられていた。ところが、最近の分子系統解析により、六脚類は多足類ではなく甲殻類と近縁であることが明らかになった。さらに、ミトコンドリア遺伝子を用いた研究では無翅昆虫の内顎類(カマアシムシ目+トビムシ目+コムシ目)は甲殻類より前に分岐した可能性が示唆され、六脚類の単系統性も疑問視されていた。しかし、我々のこれまでの研究結果7)を含め、多くの研究8)では六脚類が単系統である可能性が非常に高いことが示唆されている。
一方、各亜門における「綱」間・「目」間の系統関係は混乱しており明確になっていない。これまで統一した見解は得られていなかったが、六脚類内の「目」間の系統関係については我々の研究7,9)によって理解が大きく前進した。しかし、その六脚類でも完全変態類以外では検討の余地が残されている。地球上においてもっとも多様化している六脚類をはじめとする節足動物門の進化を理解するためには、まずこれらの生物群の系統関係を明らかにすることが不可欠であり、極めて重要な課題である。多くの研究が行われてきたにもかかわらず十分な理解が進まないもっとも大きな原因は、解析に使用された分子情報の不適切性と情報量の不足であると考えられる。そこで、我々は六脚類あるいは節足動物門全体の系統解析に適切であると思われる核ゲノム上のタンパクをコードしている複数の遺伝子を比較して、六脚類特に昆虫類をはじめとする節足動物門の系統進化の解明を目指してきた。
このような背景のもと、今年度は主として多足類の系統解析の結果について報告する。
結果と考察
1)多足類の系統解析
多足類は、ムカデ綱 Chilopoda(5目)、ヤスデ綱 Diplopoda(16目)、コムカデ綱 Symphyla(1目)、エダヒゲムシ綱 Pauropoda(2目)の4綱からなる。多足類と鋏脚類との系統関係については、両者が姉妹群を形成するMyriochelata説と、多足類が汎甲殻類(甲殻類+六脚類)と姉妹群を作る大顎類 (Mandibulata) 説が提唱されている。近年の分子系統学の研究では大顎類説がより支持を集めている。また、多足類の単系統性と各綱の単系統性についてもおおむね支持されている。
一方、多足類内の綱レベルの系統関係については、分子系統解析の結果8,10)からコムカデ綱とエダヒゲムシ綱が姉妹群を形成するという説 (Edafopoda説) が示されている。しかし1)コムカデ綱とエダヒゲムシ綱には共有派生形質(単系統性を支持する形態的特徴)が知られていないこと、2)各系統群の支持率が低いこと、3)サンプリングが充分でないことなどの問題点がある。また、ムカデ綱とヤスデ綱内の目レベルの系統関係についても一致した結論が得られていない。
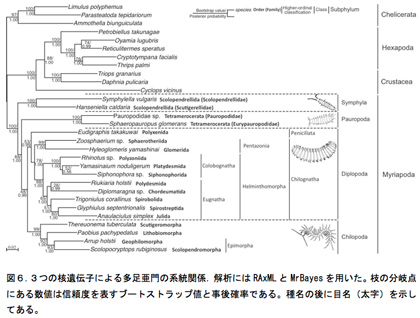
本研究では核DNAにコードされた3種のタンパク質遺伝子RNA polymerase II largest subunit (RPB1), RNA polymerase II second largest subunit (RPB2) とDNA polymerase δ catalytic subunit (DPD1)のアミノ酸配列を用いて、最尤法(RAxML)とベイズ法(MrBayes)による系統解析を行った。ムカデ綱とヤスデ綱の全ての目から1種ずつ、コムカデ綱コムカデ目の2科とエダヒゲムシ綱エダヒゲムシ目の2科の各科から1種ずつ、計19種を用いて上記の3遺伝子の塩基配列を決定した。得られた配列情報をもとに推定されたアミノ酸配列を用いて系統解析を行っている。今年度は、より適切な外群の選定やLBA(Long Branch Attraction;系統樹上で長い枝を持つ系統同士が本来の系統関係に関係なく近縁であると推定される現象)の影響の除去など、信頼性の高い系統関係を推定するための解析作業を行った。その結果、図6に示した系統樹が最終的に得られた。
綱レベルの系統関係についてはコムカデ綱が最初に分岐し残り3綱(ムカデ綱・ヤスデ綱・エダヒゲムシ綱)がー系統群であることが示唆された(図6)。この結果はこれまでに提唱されてきた多くの説と異なっている。コムカデ綱が六脚類に近縁でありムカデ綱・ヤスデ綱・エダヒゲムシ綱の3綱が単系統群をなすこと(Monomalata説)を顎の構造に基づいて1960年代にSharovが唯一示唆している11)。コムカデ綱と六脚類との近縁性を支持しない点でSharovの考え方と完全には一致しないが、我々の結果はMonomalata説とは矛盾しない。逆に言うと、我々の結果は顎の構造からも支持されていることになる。
ムカデ綱とヤスデ綱内の目レベルの系統関係については形態に基づくこれまでの仮説と矛盾しない結果になった。ヤスデ鋼内では唇顎類(Chilognatha)・前雄類(Helminthomorpha)・畸顎類(Colobognatha)の3分類群の単系統性が強く支持される結果が得られた(図6, 2012年度活動報告)。
2)多足類の分岐年代の推定
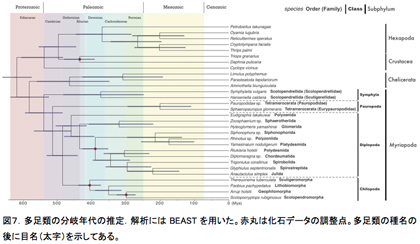
多足類の化石情報から、多足類の最初の分岐(ムカデ綱とその他の多足類の分岐)はシルル紀中期かそれ以前であると考えられている。一方、近年の分子系統解析に基づく推定ではその分岐がより古いカンブリア紀後期に遡ることが示唆されている。しかし、我々の系統解析の結果では、最初に分岐した多足類はムカデ綱ではなくコムカデ綱である可能性が高いことが示唆された(1参照)。この結果は、実はカンブリア紀後期よりもさらに古い時代に多足類の分岐が始まっていたことを示唆している。そこでBEASTを用いて、4つの化石情報と系統解析に使用した配列情報に基づいた多足類の分岐年代推定を行った。その結果、多足類の綱間の分岐が起きたのはカンブリア紀初期からオルドビス紀初期と推定された(図7)。この結果は、多足類の最初の分岐(コムカデ綱とその他の多足類との分岐)がカンブリア紀初期に遡ることを意味している。カンブリア紀には土も陸上生物も存在していなかったと考えられている。我々の結果は、節足動物門においてこれまで考えているよりももっと多くの系統群が陸上への適応を独立に果たした可能性があることを示唆している。
各綱内では、ムカデ綱の目レベルの分岐はデボン紀からペルム紀初期までの間に起きたことが示唆された。一方ヤスデ綱の目レベルでは分岐年代の幅がより大きかった。最初の分岐はオルドビス紀にまで遡り、最も近縁な2目の分岐は中世代の中期であると推定された(図7)。コムカデ綱2科の分岐が少なくとも古生代にまで遡り、ムカデ綱とヤスデ綱の一部の目間の分岐よりも古いことが示唆された(図7)。興味深いことに、コムカデ綱は多足類において最初に分岐したにもかかわらず、形態分類学的には1目2科しか認められていない。つまりコムカデ綱の形態の多様化は他の多足類と比べると相当低いことになる。これらのことから、コムカデ綱は数億年(少なくとも2億5千万年、図7参照)の間にあまり形態変化を起こしていない可能性もしくは多様化したけれど2科を残して絶滅した可能性が考えられる。
3)多足類の祖先形質の推定
多足類は、その名前からも分かるように脚の多い動物で、多い場合は数百対もの脚をもつ。また、多足類の脚は各体節に付いているので、脚が多いことは体節数も多いことを意味する(ちなみに、ヤスデ綱には各体節に2対の脚があるのに対してその他の多足類は各体節に1対の脚だけがある)。
大部分の多足類は孵化した後に成長しながら体節(と脚)を増やしていく(増節)。このような変態様式は脱皮と増節の起こり方によって4型(真増節変態, Euanamorphosis・半増節変態, Hemianamorphosis・完増節変態, Teloanamorphosis・整形変態, Epimorphosis)に分けられている。真増節変態では一生にわたって脱皮に伴い増節が起こる。半増節変態では、脱皮は一生にわたって起こるが脱皮に伴った増節はある時期(成虫になった時期?)に達した時に起こらなくなる。完増節変態では脱皮に伴った増節がある時期(成虫になった時期?)まで起こり、その後脱皮も増節も起こらなくなる。一方、整形変態では、脱皮は起こるものの増節は全く起こらない。これらの変態様式は必ずしも多足類の分類群ごとにまとまっているわけではない。それでは多足類のもともとの祖先種はどんな変態様式をしていて、いつから多足類は脚が多くなったのだろうか。多足類だけでなく多くの甲殻類・六脚類カマアシムシ目・鋏角類ウミグモ綱・鋏角類の化石種さらには三葉虫にも見られるので、半増節変態が節足動物門の祖先的様式であると考えられている。また、多足類の形態分類と形態に基づく系統関係から多足類において変態様式の進化は少なくとも2回起きたのではないかと推測されている。
「いつから多足類は脚が多くなったのか」という疑問に答えるために、Mesquiteを用い多足類の系統樹に基づく祖先形質の復元推定を行った。その結果、多足類の祖先種も半増節変態であったことが示唆された(図8)。また、半増節変態(図8黄)の現生の多足類の多くは体節数が少ない(図8)ことから、多足類の祖先種は体節数と脚の数が比較的少なかったと考えられた。
また多足類において真増節変態(図8緑)と完増節変態(図8赤)は前雄類(Helminthomorpha)で進化したことが明らかになった。これらの解析の結果から2つの対立的な進化仮説を立てることができる。一つは真増節変態と完増節変態が独立的に(真増節変態は複数回)半増節変態から進化した仮説である。もう一つは前雄類の共通祖先種の時点で半増節変態から真増節変態に変わり、その後前雄類のある系統では完増節変態が進化し、ある系統で再び半増節変態に先祖返りした仮説である(図8)。
![図8.多足類の祖先形質(変態様式)の推定.青、黄、赤、緑の4色は4種類の変態様式(本文参照)を表す。図の左側にある系統樹の分岐点にある○の色は変態様式の割合を示す。図の右側に、種名、目名(科名)、[体節数]、上位分類群名という順に示す。](http://www.brh.co.jp/research/lab02/activity/img/13/img_figure13.jpg)
4)ゲノムDNAから多足類の系統解析分子マーカー配列の取得
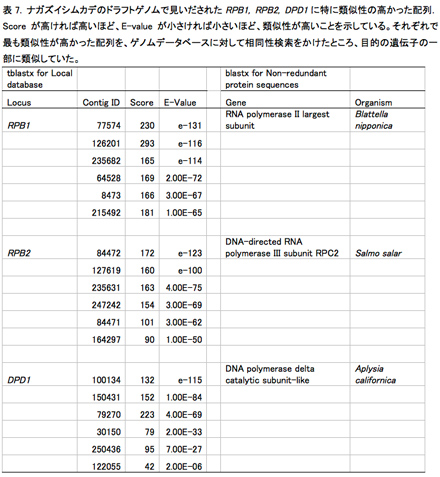
これまで多足類について、核DNAにコードされた3種のタンパク質遺伝子RPB1, RPB2とDPD1のアミノ酸配列を用いて系統解析を行ってきた(1参照)。これらの配列情報の取得には新鮮な生体試料RNAから合成したcDNAが必要である。ところが、ムカデ綱の目レベルの系統関係を明らかにするために新規に解析に加える必要のあったナガズイシムカデ目(以下、ナガズイシムカデ)は日本に分布しておらず、生体試料を入手することができなかった。また、ゲノム情報も明らかでなく、ナガズイシムカデのRPB1, RPB2とDPD1の塩基配列について参考にできる情報がない状態だった。そこで、ナガズイシムカデのゲノムDNAを次世代シーケンサで解析することにより、ナガズイシムカデのRPB1, RPB2とDPD1をコードする塩基配列の決定を試みた。無作為に断片化したゲノムDNAから構築したライブラリーを用い、2ラン分の情報を取得した。その結果、およそ3300万の塩基配列が得られた。CLC Genomic Workbenchを用いて、これらの配列のアセンブルを行った結果、最大で18,000塩基のコンティグ配列が31万個得られた(表6)。これら440して、tblastxを用いて近縁種のイシムカデPaobius pachypedatus の配列をもとに相同性検索を行ったところ、断片的ではあるがRPB1, RPB2とDPD1すべての配列が見いだされた(表7)。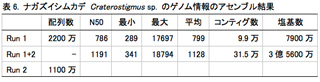
今後、これらの配列情報をもとに、それぞれの遺伝子の完全長の塩基配列決定を試みる予定である。今回の試みによって、ゲノム情報のない種であっても、ドラフトゲノム配列(完全ではないゲノム情報)さえあれば、新規の分子マーカー配列が得られる可能性が示唆された。しかし、得られた情報のうち用いるのはほんの一部で、残りの大部分は実質無駄な情報となってしまう。そのため、今後、特定の塩基配列を濃縮するライブラリー作成法など、より効率のいい方法を検討する予定である。
おわりに
本研究の結果による大きな発見は、多足類においてコムカデ綱が最初に分岐することが始めて示されたことである。これまで、多足類の綱レベルの系統関係についてさまざまな議論がなされており多くの仮説が提唱されてきた。しかし、決定的な証拠が得られておらず結論には至っていない。本研究の結果はこの状況に一石を投じるに違いない。今後、ヤスデ綱の材料を増やしすべての目を用いた目レベルの系統関係を解明したい。さらに鋏角類と汎甲殻類の情報も加えて節足動物門全体の系統解析を行い、その進化を探りたい。また、甲殻類の材料を充実させることで、六脚類に最も近縁な甲殻類が鰓脚類であるかを解明し、そして最終的には六脚類の起源を明らかにしたい。
引用文献
- 1) Herre E.A., Jander K.C. and Machado C.A. (2008) Evolutionary ecology of figs and their associates: recent progress and outstanding puzzles. Ann. Rev. Ecol. Evol. Syst. 39: 439-458.
- 2) Su Z.-H., Iino H., Nakamura K. Serrato A. and Oyama K. (2008) Breakdown of the one-to-one rule in Mexican fig-wasp associations inferred by molecular phylogenetic analysis. Symbiosis 45: 73-81.
- 3) Cornille, A., Underhill, J.G., Gruaud, A., Hossaert-McKey, M., Johnson, S.D., Tolley, K.A., Kjellberg, F., von Noort, S. and Proffit, M. (2012) Floral volatiles, pollinator sharing and diversification in the fig-wasp mutualism: insights from Ficus natalensis, and its two wasp pollinators (South Africa). Proc. R. Soc. B 279: 1731-1739.
- 4) Cruaud et al. (2012) An extreme case of plant–insect codiversification: figs and fig-Pollinating wasps. Syst. Biol. 61: 1029-1047.
- 5) Azuma H., Harrison R.D., Nakamura K. and Su Z.-H. (2010) Molecular phylogenies of figs and fig-pollinating wasps in the Ryukyu and Bonin (Ogasawara) islands, Japan. Genes Genet. Syst. 85: 177-192.
- 6) Kusumi, J., Azuma, H., Tzeng, H.-Y., Chou, L.-S., Peng, Y.-Q., Nakamura, K., and Su, Z.-H. (2012) Phylogenetic analyses suggest a hybrid origin of the figs (Moraceae: Ficus) that are endemic to the Ogasawara (Bonin) Islands, Japan. Mol. Phylogenet. Evol. 63: 168-179.
- 7) Sasaki, G., Ishiwata, K., Machida, R., Miyata, T. and Su, Z.-H. (2013) Molecular phylogenetic analyses support the monophyly of Hexapoda and suggest the paraphyly of Entognatha. BMC Evol. Biol. 13:236.
- 8) Regier J.C., Shultz J.W., Zwick A., Hussey A., Ball B., Wetzer R., Martin J.W., Cunningham C.W. (2010) Arthropod relationships revealed by phylogenomic analysis of nuclear protein-coding sequences. Nature 463: 1079 –1083.
- 9) Ishiwata K., Sasaki G., Ogawa G., Miyata T. and Su Z.-H. (2011) Phylogenetic relationships among insect orders based on three nuclear protein-coding gene sequences. Mol. Phylogenet. Evol. 58: 169-180.
- 10) Dong Y., Sun H., Guo H., Pan D., Qian C., Hao S., Zhou K. (2012) The complete mitochondrial genome of Pauropus longiramus (Myriapoda: Pauropoda): Implications on early diversification of the myriapods revealed from comparative analysis. Gene 505: 57–65.
- 11) Sharov A.G. (1966) Basic Arthropodan Stock with special reference to insect. Pergamon Press, Oxford.
