年度別活動報告
年度別活動報告書:2012年度
分子系統から生物進化を探る 3-2. 六脚類の起源と節足動物の系統進化
蘇 智慧(主任研究員) 岡本朋子(奨励研究員)
佐々木綾子(研究補助員) 宮澤秀幸(大阪大学大学院生)
はじめに
現生の節足動物は、記載種数において動物界最大の分類群であり、鋏角亜門 (Chelicerata)、多足亜門 (Myriapoda)、甲殻亜門 (Crustacea)と六脚亜門 (Hexapoda) の4亜門に分類され、節足動物門をなす。この節足動物門に属する生物は、地球上の至る所に分布しており、人類にとっても身近なものが多い。しかし、節足動物門内の系統関係は未だ明確になっていない点が多い。従来、鋏角類は節足動物門の中で祖先的な位置にあり、六脚類は多足類と近縁であると考えられていたが、最近の分子系統解析により、六脚類は多足類ではなく甲殻類と近縁であることが明らかになった。さらに、ミトコンドリア遺伝子を用いた研究では、無翅昆虫の内顎類(カマアシムシ目+トビムシ目+コムシ目)は甲殻類より前に分岐した可能性が示唆され、六脚類の単系統性にも疑問がもたれた。しかし、これまでの我々の研究結果では六脚類が単系統である可能性が非常に高いことが判明した。一方、各亜門における「綱」間、「目」間の系統関係は混乱しており、明確になっていない。六脚類内部の「目」間の系統関係については、様々な研究が行われてきたにも関わらず、明快な答が少なく、我々の研究7)によって理解が大きく前進した。しかし、完全変態類以外ではまだ不明なところが多く残っているのも事実である。地球上においてもっとも多様化している昆虫類を始め、節足動物の進化を理解するには、これらの生物群の系統関係を明らかにするのが基礎であり、極めて重要である。昆虫類を始め、節足動物の系統関係について、これまで多くの研究が行われてきたにもかかわらず、なぜなかなか解明できないのか。これまでの解析において使用していた分子情報の不適切性と情報量の不足がもっとも大きな原因であると考えられる。そこで、我々は六脚類或いは節足動物全体の系統解析に適切であると思われる核ゲノム上のタンパクをコードしている複数の遺伝子を比較して、昆虫類をはじめとする節足動物の系統進化の解明を試みている。今年度は主として多足類と鰓脚類(甲殻亜門)の系統解析の結果について報告する。
結果と考察
1)多足亜門の系統関係
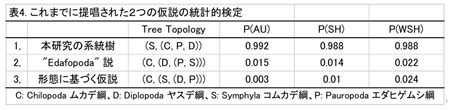
多足亜門は、ムカデ綱(5目)、ヤスデ綱(16目)、コムカデ綱(1目)、エダヒゲムシ綱(2目)の4綱からなる。多足亜門と鋏脚亜門との関係については、両者が姉妹群を形成するMyriochelata説と、多足亜門が汎甲殻類と姉妹群を作る大顎類 (Mandibulata) 説が提唱されている。近年の分子系統学の研究により、節足動物門における多足亜門の単系統性や各綱の単系統性についてはおおむね支持されている。多足亜門の4綱間の系統関係については、分子系統解析の結果8,9)から、エダヒゲムシ綱とコムカデ綱が姉妹群を形成するという説 (Edafopoda説) が示されているが、エダヒゲムシとコムカデには共有派生形質が知られておらず、またこの解析結果についても、その支持率が低いことや、サンプリングが充分でないことなどの問題点があり、その結論は明瞭でない。また、ヤスデ綱とムカデ綱内部の目レベルの系統関係については、分子系統解析から一致した結論が得られていない。
本研究では核DNAにコードされた3種のタンパク質遺伝子RNA polymerase II largest subunit(RPB1), RNApolymerase II secondlargest subunit (RPB2) とDNA polymerase δ catalytic subunit (DPD1)のアミノ酸配列を用いて、最尤法(RAxML)とベイズ法(MrBayes)による系統解析を行った。材料としては、ムカデ綱の4目(ゲジ目、イシムカデ目、オオムカデ目、ジムカデ目)とヤスデ綱の11目(フサヤスデ目、ネッタイタマヤスデ目、タマヤスデ目、ジヤスデ目、ツムギヤスデ目、ヒラタヤスデ目、ツムギヤスデ目、オビヤスデ目、ヒメヤスデ目、フトマルヤスデ目、ヒキツリヤスデ目)のそれぞれの目から1種ずつ、コムカデ綱コムカデ目の2科(ナミコムカデ科、ヤサカコムカデ科)とエダヒゲムシ綱エダヒゲムシ目の2科(エダヒゲムシ科、ヨロイエダヒゲムシ科)の各科から1種ずつ、計19種を用いて上記の3遺伝子の塩基配列を決定し、これをもとに推定されたアミノ酸配列を用いて系統解析を行った。また、CONSELを用いて、本研究で得られたデータセットに基づき、他のいくつかの仮説の可能性について統計的検定を行った。
その結果、4綱の系統関係については、Edafopoda説(エダヒゲムシ綱とコムカデ綱が姉妹群を形成する)や形態分類によるProgoneata説(コムカデ綱、エダヒゲムシ綱とヤスデ綱が単系統群を形成する)などとは異なり、コムカデ綱が最初に分岐し、残り3綱(ムカデ綱、エダヒゲムシ綱、ヤスデ綱)がクレードを形成するという結果が得られた(図9)。
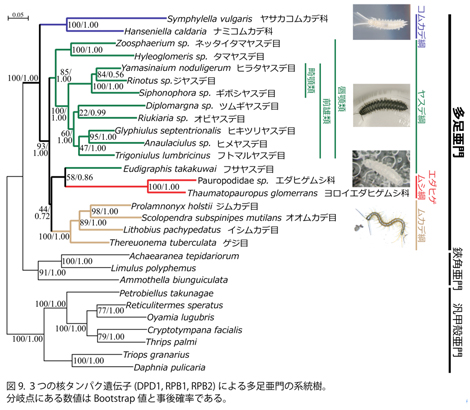
この結果は、顎の構造に基づいて、ムカデ綱、エダヒゲムシ綱、ヤスデ綱は単系統群 Monomalata をなすとするSharov 10)の仮説と一致する。また、CONSELを用いて、これまでに唱えられてきたEdafopoda説と形態に基づく仮説について統計的検定を行ったところ、これらの仮説はいずれの方法でも5%水準で棄却された(表4)。
ムカデ綱とヤスデ綱内部の系統関係については、形態に基づく仮説と一致し、ヤスデ鋼内部では、唇顎類、前雄類と畸顎類の3分類群の単系統性が強く支持される結果が得られた(図9)。
2)鰓脚類の系統関係
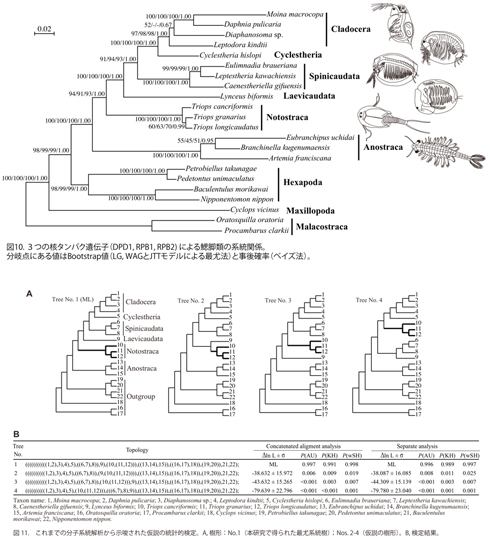
六脚類は甲殻類と共通祖先を有することが明らかになったが、甲殻類のどの系統から六脚類が分岐したのはまだ解明されていない。甲殻類は鰓脚綱Branchiopoda、カシラエビ綱Cephalocarida、ムカデエビ綱Remipedia、顎脚綱Maxillopodaと軟甲綱Malacostracaに分類されている。その中では、鰓脚類が六脚類に最も近縁である分類群の一つであることが分子情報から示唆されており、六脚類の起源を知るには鰓脚類の系統進化を解明する必要がある。
鰓脚類は分類上無甲目(ホウネンエビ目)Anostraca、背甲目(カブトエビ目)Notostracaと双殻目(ミジンコ目)Diplostracaに分けられている。双殻目はさらにタマカイエビ亜目Laevicaudata、カイエビ亜目Spinicaudata、キクレステリア亜目Cyclestheridaと枝角亜目(ミジンコ亜目)Cladoceraの4亜目に分類されている。鰓脚類の高次分類群間の系統関係について、分子と形態による多くの研究(文献)が行われてきたが、分子系統の結果は明らかでない 11-14)。我々はRPB1、RPB2とDPD13つの遺伝子(約3400aa)を用いて鰓脚類の目・亜目間の系統関係の解明を行ってきた。しかし、キクレステリア亜目は日本に生息しておらず、材料の入手は困難であった。今回、琵琶湖県立博物館の楠岡博士を通してマレーシアから材料を得ることができた。これで鰓脚類の全目・亜目のサンプルが揃うことになった。キクレステリアの上記の3遺伝子の塩基配列を決定し、アミノ酸配列に翻訳してこれまでに得られたデータセットに加え、系統解析を行った。まず、鋏角類と多足類を外群にして、他の甲殻類(軟甲類と顎脚類)や六脚類(内顎類と外顎類の種を含む)を加えて、系統解析を行った結果、鰓脚類が単一起源であることが確認された。鰓脚類内部の系統関係では、無甲目が最初に分岐し、その後、背甲目、タマカイエビ亜目とカイエビ亜目が順番に分かれ、最後に分岐したのはキクレステリア亜目と枝角亜目であることが判明した(図10)。この系統関係は統計的に有意に支持され、信頼性が極めて高いものである。この結果は形態から推定された系統関係ともほぼ一致し、双殻目の単系統性も支持された(図6)14)。さらに、これまでの分子系統解析の研究から提唱された複数の仮説について、CONSELを用いて統計的な検定を行った。その結果、それらの仮説はすべて1%水準で棄却された(図11)。
おわりに
本研究の結果により、多足亜門においてコムカデ綱が最初に分岐することが始めて示され、大きな発見である。多足亜門の系統関係の研究では、4綱の関係についてさまざまな議論がなされており、多くの仮説が提唱されてきた。しかし、決定的な証拠が得られておらず、結論には至っていない。本研究の結果は大きなインパクトを与えることに違いない。今後、ヤスデ綱の材料を増やして全目の目間の関係を解明し、鋏角類と汎甲殻類のデータも加えて節足動物全体の系統解析を行い、その進化を探りたい。また、六脚類の起源と関わる鰓脚類の系統関係に関しては、すべての目と亜目を含めた高次分類群間の系統解析を完成し、それら目と亜目間の系統関係が明らかになった。今後、鰓脚類が六脚類の祖先に当たる系統であるかを明らかにしていきたい。
引用文献
- 1) Herre E.A., Jander K.C. and Machado C.A. (2008) Evolutionary ecology of figs and their associates: recent progress and outstanding puzzles. Ann. Rev. Ecol. Evol. Syst. 39: 439-458.
- 2) Su Z.-H., Iino H., Nakamura K. Serrato A. and Oyama K. (2008) Breakdown of the one-to-one rule in Mexican fig-wasp associations inferred by molecular phylogenetic analysis. Symbiosis 45: 73-81.
- 3) Cornille, A., Underhill, J.G., Gruaud, A., Hossaert-McKey, M., Johnson, S.D., Tolley, K.A., Kjellberg, F., von Noort, S. and Proffit, M. (2012) Floral volatiles, pollinator sharing and diversification in the fig-wasp mutualism: insights from Ficus natalensis, and its two wasp pollinators (South Africa). Proc. R. Soc. B 279: 1731-1739.
- 4) Azuma H., Harrison R.D., Nakamura K. and Su Z.-H. (2010) Molecular phylogenies of figs and fig-pollinating wasps in the Ryukyu and Bonin (Ogasawara) islands, Japan. Genes Genet. Syst. 85: 177-192.
- 5) Kusumi, J., Azuma, H., Tzeng, H.-Y., Chou, L.-S., Peng, Y.-Q., Nakamura, K., and Su, Z.-H. (2012) Phylogenetic analyses suggest a hybrid origin of the figs (Moraceae: Ficus) that are endemic to the Ogasawara (Bonin) Islands, Japan. Mol. Phylogenet. Evol. 63: 168-179.
- 6) 岡本朋子(2012)花の匂いの捕集方法。「種間関係の生物学 〜共生・寄生・捕食の新しい姿〜」 種生物学会編、文一総合出版、P317-329。
- 7) Ishiwata K., Sasaki G., Ogawa G., Miyata T. and Su Z.-H. (2011) Phylogenetic relationships among insect orders based on three nuclear protein-coding gene sequences. Mol. Phylogenet. Evol. 58: 169-180.
- 8) Regier J.C., Shultz J.W., Zwick A., Hussey A., Ball B., Wetzer R., Martin J.W., Cunningham C.W. (2010) Arthropod relationships revealed by phylogenomic analysis of nuclear protein-coding sequences. Nature 463: 1079 –1083.
- 9) Dong Y., Sun H., Guo H., Pan D., Qian C., Hao S., Zhou K. (2012) The complete mitochondrial genome of Pauropus longiramus (Myriapoda: Pauropoda): Implications on early diversification of the myriapods revealed from comparative analysis. Gene 505: 57–65.
- 10) Sharov A.G. (1966) Basic Arthropodan Stock with special reference to insect. Pergamon Press, Oxford.
- 11) Braband A., Richter S., Hiesel R. and Scholtz G. (2002) Phylogenetic relationships within the Phyllopoda (Crustacea, Branchiopoda) based on mitochondrial and nuclear markers. Mol. Phylogenet. Evol. 25: 229–244.
- 12) Stenderup J.T., Olesen J. and Glenner H. (2006) Molecular phylogeny of the Branchiopoda (Crustacea)-multiple approaches suggest a 'diplostracan' ancestry of the Notostraca. Mol. Phylogenet. Evol. 41: 182–194.
- 13) DeWaard, J.R., Sacherova, V., Cristescu, M.E.A., Remigio, E.A., Crease, T.J., Hebert, P.D.N. (2006) Probing the relationships of the branchiopod crustaceans. Mol. Phy. Evol. 39: 491-502.
- 14) Richter, S., Olesen, J., and Wheeler, W. C. (2007) Phylogeny of Branchiopoda (Crustacea) based on a combined analysis of morphological data and six molecular loci. Cladistics 23: 301-336.
