年度別活動報告
年度別活動報告書:2013年度
アゲハチョウの食草選択と進化
尾崎 克久(研究員) 吉澤 靖貴(奨励研究員)
廣嵜 由利恵(研究補助員)
はじめに
動物は、外界に存在する化合物を味覚・嗅覚として認識している。陸棲生物においては、水溶性・脂溶性の不揮発性化合物を感覚器官が接触して認識するものを味覚、揮発性化合物を非接触的に認識するものを嗅覚と呼ぶ。化学感覚(味覚・嗅覚)は、生物間のコミュニケーションにとって不可欠な機能であり、寄主選択・配偶行動・集団生活の維持・社会性の構築など、生存上の様々な役割を担っている。寄主選択に関わる化学受容の仕組みに変化が生じた場合、それまでとは異なる空間を生息の場として利用する集団が現れ棲み分けが起こり、何世代にもわたって棲み分けが繰り返されることによって種分化へとつながると考えられている。このような変化は、進化の歴史を物語る証拠としてゲノムに刻まれる。動物と環境との関わりにおいて中心的な機能である味覚や嗅覚といった化学受容に関わる分子機構の解明は、多様化・種分化・環境への適応の仕組みを解明するために、最も有力な手がかりになると考えられる。
アゲハチョウの仲間は、他の多くの植食性昆虫と同様に、特定の植物のみを餌として利用する範囲の狭い寄主選択をしており、寄主選択とアゲハチョウ科の進化には相関関係が認められる1。卵から孵化したばかりのアゲハチョウの幼虫は、体が小さく移動能力が低いため、広い環境中を歩き回って自力で餌を探索することは困難である。そのため、飛ぶことができ移動能力の高いメス成虫による正確な植物種の識別と産卵場所の選択は、次世代の生存に決定的な影響を与えることになる。メス成虫は産卵の直前に前脚で植物に触れることで、前脚ふ節にある化学感覚子を通じて含有する化合物を感じ取り、その組み合わせによって産卵行動が引き起こされる。前肢ふ節の感覚子数は雌雄間で大きな差があり、圧倒的にメスに多く、メスのみが行う行動、つまり産卵において重要な役割を持つことが知られている。ナミアゲハ(Papilio xuthus)では、主な食草であるウンシュウミカンの葉から産卵刺激物質として10種類の化合物が単離されている2。他にも数種のアゲハ類で産卵刺激物質が明らかにされており、これら化合物の構造についてアゲハ種間で比較すると、類似性が認められる。このことから、植物の系統的近縁性とは無関係に、植物に含まれている化合物の類似性が寄主転換の可能性を支え、食性の進化を可能にしたのではないかと考えられている3。これまでに報告されている産卵刺激物質は全て不揮発性で、前脚ふ節で接触して感じていることから、アゲハチョウは前脚で「味」として認識していると考えられている。前脚での味の感じ方に変化が生じた場合、それまでとは異なる植物を選択する集団が現れて、住み分けによる隔離を出発点とする同所的種分化という現象を引き起こしたと考えられる。
ミカン科食性のアゲハチョウ間であっても、種ごとに産卵刺激物質として認識する化合物の組み合わせが異なるため、種ごとに特徴的な多様な味覚受容体を持っていることが予測される。また、味覚受容体が化合物を認識するためには、化合物が化学感覚子内のリンパ液を通り抜けて受容体に到達する必要があるため、化合物を結合して運搬する役割を持つタンパクが必要になると考えられる。味覚受容体と化合物結合タンパクを中心とする産卵刺激物質受容システムに関わる遺伝子群を解明し、複数種間で比較することができれば、食草転換を原動力として起きた進化という現象のメカニズムを理解する重要な手がかりになると考えた。
昆虫の味覚に関する研究が本格的に始まったのは30年近く前に遡るが4、味覚受容体は2000年になって初めて7回膜貫通型受容体(以下7TMRと略)が報告された5。化学受容の7TMRは一次構造の多様性が高く、脊椎動物から報告されている味覚・嗅覚の7TMRに対する類似性を手がかりとした探索は困難であり、ショウジョウバエの全ゲノム配列の情報科学的解析によって候補遺伝子ファミリーが同定された。これまでに7種の昆虫の全ゲノムが解読されているが、味覚受容体遺伝子はゲノムからコンピュータで解析されたものに限られている。興味深いことに、これまでに報告されている昆虫化学感覚受容体は、昆虫間でも類似性に乏しく、それぞれの生活史に合わせて独自の多様化をしている可能性が示唆される。昆虫の味覚受容体は極端に発現量が少ないため解析は困難を極め、機能が解明されているものはまだ少ない6,7。化合物結合タンパクについて、味覚器官で発現するものが見つかっており8、食草の選択に重要な役割を持つものも報告されている9。ナミアゲハでは、Chemosensory protein (CSP) がゲノムの特定の領域にクラスターしており10、カイコとシンテニーがある事を確認している。また、多種昆虫から化合物結合タンパクの遺伝子が報告され、情報が蓄積しつつある。
本研究は、主たる食草の産卵刺激物質が明らかにされているアゲハチョウ科昆虫を材料として用い、メス成虫前脚ふ節に発現する味覚受容体遺伝子及び化合物結合タンパク遺伝子をクローニングし、その機能と特徴の解明を目的として取り組んでいる。
昨年度までの取り組みで、ナミアゲハからメス成虫前脚ふ節に発現の特異性を示す7回膜貫通型タンパク質遺伝子(PxutGr1)を発見し、バキュロウイルス発現系を用いたカルシウムイメージング法により産卵刺激物質の一つであるSynephrine(シネフリン)に特異的に応答する受容体である事を解明している。また、RNAi法を用いた機能阻害と電気整理実験・産卵行動実験に取り組んだ結果、PxutGr1の発現を阻害したナミアゲハでは、Synephrineに対する感度が低下し、産卵行動が抑制されることを確認している。これらの結果から、ゲノムが読まれていない昆虫から初めて味覚受容体遺伝子を同定し、産卵という本能行動に関与していることを明らかにした11。また、電気生理実験により、産卵行動を引き起こすために複数の産卵刺激物質を同時に認識する必要がある仕組みについて、神経プロファイルを解明している12。
今年度は、当館に導入した次世代型シークエンサーMiSeqの、300塩基ペアエンドというロングリード性能を最大限に活用することで、ホモロジーをベースとした解析のみでは検出が難しい昆虫化学感覚受容体遺伝子について、高効率に全長配列を検出できることを確認した。
結果と考察
MiSeq活用のためのRNA-seqプロトコール改変
昨年度は、当館に導入されたばかりの次世代型シーケンサー MiSeq を用い、RNA-seq によるナミアゲハのメス成虫前脚で発現する化学感覚受容体遺伝子の探索を行った。illumine 社の TrueSeq RNA Preparation kit を用い、メーカーのプロトコールに従ってシーケンスライブラリーを作成し、MiSeq で塩基配列を決定し、約3000万本のリードが得られた。Trinity13という発現産物の配列データ結合用ソフトを用いてアセンブルした結果、ナミアゲハから約60万個の contig (結合した塩基配列の塊)が得られた。平均 contig 長は約600塩基という短さであったため、遺伝子の完全長がひとつのcontigに含まれているものは極わずかで、シネフリン受容体遺伝子PxutGr1(コード領域が1224塩基の遺伝子)も3つのcontigに分断されており、その3 contigを手作業でつなぎ合わせても完全長にはならなかった。ホモロジー検索により昆虫化学感覚受容体遺伝子にヒットした12個のcontigについて、RACE法による全長配列の取得を試みたが、PxutGr1の発見当時と同様に5'-RACEでは特定の場所で逆転写反応が止まってしまう現象が観察され、どれも成功しなかった。
今年度は、MiSeqの高出力性能とロングリード性能を活用することで、標準的な実験手法では完全長の取得が難しい場合が多い昆虫味覚受容体遺伝子の検出効率改善を試みた。RNA-seq用のライブラリーを作成するキットは数社から購入可能であるが、illumine 社の TrueSeq RNA Preparation kit V2 では逆転写反応の前にmRNAを切断し、ランダムプライマーを用いて逆転写反応を行い、ショットガンシークエンスのような要領で配列を決定する手法であるため、前述の逆転写反応が途中で止まってしまうことによる問題を克服できると思われるので、インサートサイズを長くする工夫をすることでアセンブルの効率を改善できるのではないかと考えた。
標準プロトコールのmRNA切断作業では、
94℃ 8分 → 4℃ ホールド
というプログラムになるが、この方法ではインサートサイズが短くなってしまう(図1A)。切断反応が進行するのは94℃に保温している間なので、この時間を短縮するとインサートサイズを長くすることが可能である事を確認した(図1B)。
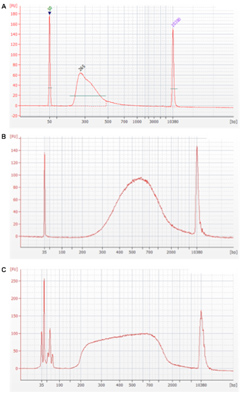
図1 mRNA切断行程における保温する時間とインサートサイズの関係
A: 94℃に8分間保温した場合 B: 94℃に加温する作業を省略した場合 C: 70℃で2分間保温した場合
保温時間とおおよそのインサートサイズの一覧を、表1にまとめる。
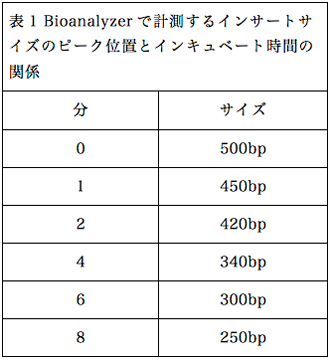
保温時間0分は、試薬類を混合した状態で加温せずに、そのまま次の行程に作業を進めるという手順であるが、最終的なライブラリー収量が加温した場合と比べて1/4〜1/8に減少する傾向があるため、実験に使用できるRNA量がふんだんにある場合のみに行うのが無難である。実験に用いるtotal RNA量が1µg以下で長鎖インサートのライブラリーを作成する場合は、94℃に30〜60秒程度加温すると良いことを確認した。
また、切断反応の温度を下げることでもインサートサイズが変化するが、作成したライブラリーのピークパターンがブロード(幅広)になる傾向が強い(図1C)。様々な長さのリードが混在することで、アセンブルの効率が上がる可能性にも期待できるため、実験の目的に合わせて使い分けることでMiSeqをより有効に活用できると考えられる。
ただし、実際にMiSeqで塩基配列を決定すると、低分子の配列が優先的に読まれる傾向が強いため、出力された配列データに含まれる長鎖リードの数が極わずかである場合が多い。長鎖リードのみを使って、contigサイズが大きくなるアセンブルを行いたい場合は、アガロースゲルを用いたサイズセレクションを行うことが効果的である。その際、アガロースゲルからマーカーレーンの部分を切り取って染色し、定規を当てて写真を撮影し、定規の目盛りを参照してサンプルを切り取ることで、UV照射によるダメージを与えずにライブラリーのサイズセレクションを行うことが可能である(図2)。
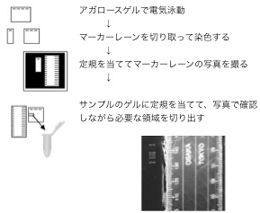
図2 アガロースゲルを用いてサイズセレクションを行う際、サンプルにUVを照射せずに切り出す手順
RNA-seqによる組織間の発現量解析
前述の結果を踏まえ、ナミアゲハのメス成虫の前脚と触角からtotal RNAを抽出し、それぞれ1µg使用してRNA-seqライブラリーを作成した。mRNAの切断反応は94℃で1分間とし、約400bp〜600bpの範囲でアガロースゲルを用いたサイズセレクションを行い、MiSeq Reagent Kit V3 600 cyclesを使ってマルチプレックスで配列を決定した。
前脚・触角ともに約2600万本のリードが得られ、リードの平均長は296bpとなり、250bp未満のリードは0.8%しか含まれていなかった。Trinityを用いたアセンブルでそれぞれ8万個と9万個のcontigが得られ、どちらも平均長は約930bpであった。標準プロトコールのRNA-seqでは、約60万個のcontigができ、平均長が600bpであったので、プロトコールの変更によって大きな改善があったと判断できる。
構築したcontigに元のリードをマッピングし、2組織間で発現量が大きく異なるものを抽出すると(図3)、前脚で発現量が多いcontigが2437個となった。これらの配列を、BLASTによるホモロジー検索および膜貫通領域の推定により化学感覚受容体遺伝子の候補として絞り込んだ結果、PxutGr1の全長配列を含む20個のcontigが抽出された。これらのcontigは昨年度に報告した12個の部分配列を含んでおり、そのうち9個は開始コドンから終止コドンまでアミノ酸配列として約400残基連続していることから、化学感覚受容体遺伝子の全長配列であると期待できる。これらアミノ酸配列をSOSUIとTMHMMを用いて膜貫通領域を推定すると、5〜8回の膜貫通領域を有すると推定された。前脚で発現量が多いcontigから推定されたものであることから、産卵刺激物質を認識するための味覚受容体であることに期待できる。
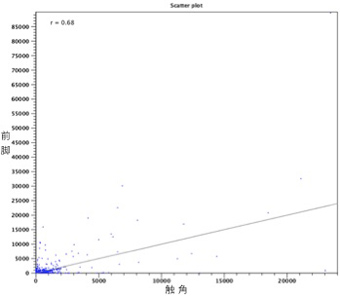
図3 組織間の発現量比較
ただし、ライブラリー作成時にサイズセレクションを行っているため、マッピングされたリード数が発現量を正確に反映していない可能性が考えられる。構築したRNA-seqライブラリーから、de novoアセンブルによって遺伝子の全長配列を取得することを目的としてサイズセレクションを行ったものと、マッピングによって発現量を推定するためにサイズセレクションを行っていないものを別にランした方が、より正確なデータになるのではないだろうか。
次世代型シークエンサーMiSeqを有効に活用することにより、発現量の低さと一次配列の多様性の高さから検出が難しかった味覚受容体遺伝子の候補配列を効率よく探索することが可能となった。昆虫化学感覚受容体遺伝子の場合、これまでは部分配列が見つかっても、通常のRACE法による全長配列の取得が困難である等の問題も見られたが、全長配列を含むと期待できるcontigが検出されるなど、RNA-seqのプロトコールの変更の効果でその問題の克服に成功した。
MiSeqによるアゲハチョウゲノム解読
MiSeqは頻繁にバージョンアップが行われ、現在では2500万クラスターを300bpのペアエンドで配列決定ができるため、1ランあたり5000万リード・16ギガベース(Gb)のスループットとなっている。平均200メガベース(Mb)のアゲハチョウゲノムであれば、1ラン当たり約80倍の重複率となるため、MiSeqだけでゲノム配列を解読することが可能になると期待できる。また、Nexteraという、改変型トランスポゾンを使って少量のゲノムDNAから簡便な操作によって短時間でライブラリーを作成できるので、組織量の少ない昆虫に好適な機器である。
Nexteraによるライブラリー作成を標準プロトコールに従って行うと、約500bpのインサートサイズになる。このライブラリーを300bpペアエンドで配列決定すると、大部分のリードがペアエンド同士でオーバーラップすることになる。ライブラリーをペアエンド間でオーバーラップしない長さで作成することが出来れば、そのペアエンド自体がメイトペアとしてscaffoldingに有効に働くことになるため、de novoアセンブルの効率を大幅に改善する。そのために、Nexteraの簡便性を損なわずに長鎖インサートのライブラリーを作成できるようにプロトコールを改変した。
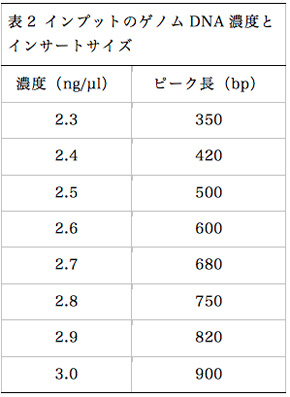
Nexteraの標準プロトコールでは、2.5ng/µlのゲノムDNA溶液を出発材料として使用するが、この濃度を変更することでライブラリーのインサートサイズをある程度任意に調節することができる(図4)。インプットのゲノムDNA濃度と、構築されるライブラリーのインサートサイズの関係を表2にまとめる。
300bpペアエンドのリードがオーバーラップしないインサートサイズのライブラリーを作成し、前述のゲルセレクション法により精製を行い、2ラン分のデータでde novoアセンブル(CLC bio Genomic Workbenchを使用)を行うと、標準プロトコールでライブラリーを作成した場合と比較してscaffold N50の値と最大scaffoldサイズの両方が大幅に改善することを確認した(表3)。ゲノム支援のアセンブルでは、全ゲノムの200倍を超えるデータ量を使用しているのに対し、今回使用したMiSeqのデータ量は大凡40倍であることを考えると、高効率にscaffoldingされていることがわかる。
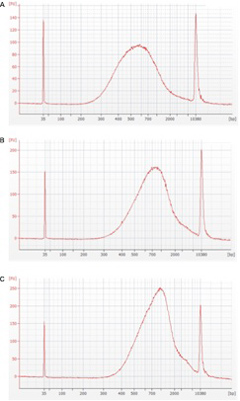
図4 Nexteraによるライブラリー作成時、インプットDNAの濃度によってインサートサイズが変化する
A: 2.6ng/µlのゲノムDNAを用いて600bpにピークがあるライブラリーを作成した
B: 2.8ng/µlのゲノムDNAを用いて750bpにピークがあるライブラリーを作成した
C: 3.0ng/µlのゲノムDNAを用いて900bpにピークがあるライブラリーを作成した
構築したナミアゲハゲノムscaffoldに、前述のRNA-seq contigをマッピングした結果、PxutGr1は約16Kbのcontigの中にその全エクソンがマップされた。他の味覚受容体遺伝子候補配列19個についても同様にマッピングした結果、同一のscaffoldに複数マップされるものは見つからなかった。この結果は、味覚受容体遺伝子がゲノム上の様々な領域に散在しており、近傍にクラスターしているのではない可能性が考えられる。しかし、化学受容体遺伝子に化合物を運搬する役割を持つのではないかと考えられているCSP遺伝子は、ゲノム上の約76Kbの領域にクラスターしていることを確認しているが10、この領域が複数のscaffoldに分断されていたことから、まだアセンブルを改善する余地が大きいことを示している。
今後、データの追加と複数サイズのメイトペア(3Kb、5Kb、10Kbを予定)の追加により、scaffoldサイズやN50の値がどこまで改善できるか確認する予定である。小型シークエンサーMiSeqを使って、数ラン分のデータ量でアゲハチョウのドラフトゲノムの作成が可能になるのであれば、MiSeqの活用の幅と研究テーマが大きく広がることに期待できる。
現在、塩基配列のリードを更に小さいk-merという単位に分断してアセンブルの計算を行うハッシュ法を利用しているが、このアルゴリズムは元々1リードあたり100bp未満のショートリードタイプのシークエンサーデータ向けに開発されたものである。計算機資源に対して小さな付加で計算できるメリットがある代わりに、大量のシークエンスデータを必要とするというデメリットがある。MiSeqの300bpペアエンドというロングリード性能を活かすために、サンガーシークエンス時代に利用されていたオーバーラップ法のアセンブラーを試してみることも有効ではないかと考えられる。しかし、オーバーラップ法のアルゴリズムはコンピューターに広大なメモリを要求するため、大型の計算機の借用が必要になる。
シークエンスライブラリーを丁寧に作成すれば、1ランあたりでメーカーのカタログスペックを上回るデータ量を取得できるが、データ中から低クオリティのリードやライブラリー作成過程で出来たPCR Duplicateを除去すると、データ量がほぼ半減してしまう。これは通常見られる現象ではあるが、より高クオリティのデータが取れるように工夫することで、削減幅を減らすことも可能ではないかと思われる。
ナミアゲハ幼虫における味覚感覚器の電気生理学的解析
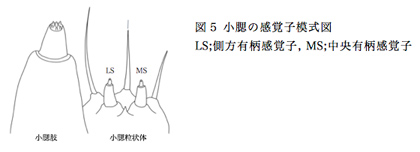
ナミアゲハ幼虫は、成虫の産卵刺激物質とは別の化合物を認識し、摂食行動が誘発される14。幼虫における食草選択機構の解明を目的とし、幼虫の主な化学感覚器官である触覚、小腮、上唇のうち味覚感覚器とされる小腮、上唇に存在する感覚子において認識する化合物の同定を試みた。今回用いた電気生理学的手法の1つであるチップレコーディング法は、感覚子を刺激する化合物溶液で満たしたガラス電極を感覚子に接触させることで、化合物が味覚受容体のリガンドであれば、その際に生じる活動電位を計測することができる。これにより感覚子ごとに認識される化合物を同定することが可能である。小腮は小腮粒状体と小腮肢の2つの器官から構成されるが、今回は小腮粒状体に存在する2本の側方および中央有柄感覚子において(図5)、いくつかの化合物に対して応答を観察することができた。
1. 糖類に対する応答
スクロース、フルクトース、グルコースの3種の糖類に対して応答する感覚子があるか調べたところ、スクロースおよびフルクトースへの応答が中央有柄感覚子にて確認された。側方有柄感覚子において応答は確認できなかった(図6)。また、少なくとも10mMの濃度においてはグルコースへの応答は中央および側方の両感覚子において確認できなかった(データ未掲載)。Murata et al (2011)らはグルコースのナミアゲハ幼虫への摂食促進効果を報告しており、別の感覚子においてグルコースを受容していることが示唆される。
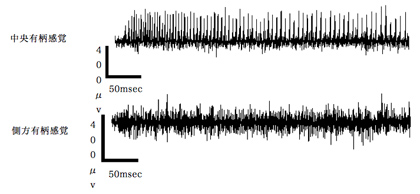
図6 スクロースの小腮粒状体上の中央有柄感覚子および側方有柄感覚子への応答
2. 苦味物質に対する応答
カフェイン、キニーネに対する応答が側方有柄感覚子において確認できた。また、キニーネにおいては側方有柄感覚子特異的に応答を引き起こすことを確認した(図7)。キニーネに対しては頻度と電位の異なる2種類の電気的応答が確認できたことから、同時に2つの味細胞が応答しているものと考えられた(図8)。成虫の産卵行動においては、重要な物質と考えられるシネフリンに対して同様に2種類の細胞が応答しており、幼虫の摂食行動では摂食刺激物質より忌避物質の認識が重要である可能性が示唆される。
3. スタキドリンに対する応答
成虫における産卵刺激物質のうち、スタキドリンは唯一接触刺激物質でもあることが報告されている14。10mMスタキドリンに対する応答は、側方および中央有柄感覚子の両方で確認された(図9)。
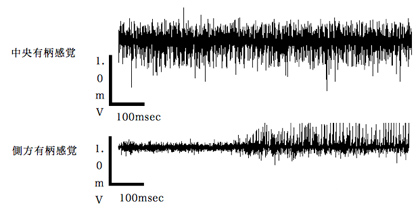
図7 キニーネの小腮粒状体上の中央有柄感覚子および側方有柄感覚子への応答
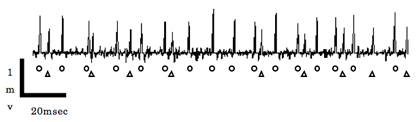
図8 キニーネの側方有柄感覚子への応答パターン。丸と三角はそれぞれ異なる細胞の興奮で生じたスパイクを示す。それぞれ0.8-1.2mVおよび0.4-0.6mV程の電位を持つ。
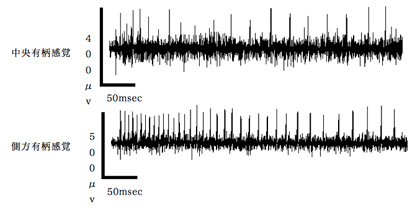
図9 スタキドリンの小腮粒状体上の中央有柄感覚子および側方有柄感覚子への応答
昨年度、成虫では3種類の産卵刺激物質認識ニューロンと、1種類の忌避ニューロンの構成によって産卵行動が制御されている可能性を報告した。今回の結果から、幼虫の摂食行動は主に糖類など一般的な物質で引き起こされ、食草選択としては忌避物質の認識が重要である可能性が示唆された。
ナミアゲハ幼虫における電気生理の手法を確立することができたので、今後これまで応答が確認できた化合物を組み合わせて、それぞれの化合物が同一の神経細胞で受容されるのか、異なる細胞で受容されるかを探っていく。
おわりに
今年度の取り組みにより、MiSeqのロングリード性能を活用し、通常の実験手法では取得が難しい昆虫味覚受容体遺伝子の塩基配列について、効率的に全長配列の取得が可能になった。RNA-seqは、解析対象となる組織で発現する遺伝子を網羅的に検出できる可能性があり、ゲノムが読まれていない非モデル昆虫の研究においては、新規遺伝子探索に有効な手段である。別組織間で発現量を比較することにより、研究対象となる組織に特異性のある遺伝子の検出を高速化する。何らかの理由でRACE法による全長配列の取得が難しい場合であっても、RNA-seqによって短期間で全長を取得可能である。この他にも、非モデル生物でRNA-seqに取り組むにあたり、MiSeqを最大限活用するための様々な工夫を行っている。これら実験手法は、羊土社実験医学別冊「次世代シークエンス解析スタンダード 実践編4『非モデル生物のRNA-seqとde novoゲノム』」にまとめた。
Nexteraを用いたゲノム解読では、ペアエンド間でオーバーラップしないインサートサイズでライブラリーを作成することにより、scaffoldingの効率が改善され、標準プロトコールで作成した場合と比較して大きなcontig/scaffoldが作成できた。MiSeqを有効に活用することで、単独の研究室でもアゲハチョウのゲノムを解読できる可能性を示した。今後数年の間に、同様の手法を用いて多くの昆虫ゲノムが解読されることが予想される。この様なドラフトゲノムをデータベース化し、情報を効率よく利用できる仕組みを構築することができれば、昆虫と植物の相互作用を解明する研究を促進するであろう。
次世代型シークエンサーの活用により、解析対象となる遺伝子の探索は大幅に高速化されているので、その機能解析のハイスループット化について取り組む必要がある。味覚受容体遺伝子の候補配列については、RNAiとチップレコーディング法を用いてリガンドを特定する計画であったが、多数ある候補遺伝子について複数のリガンド候補を使って感覚子単位で刺激を行う作業は大きな労力と長い時間を必要とする。現在、前脚ふ節全体で応答をとる「バルクレコーディング」とでもいうべき実験手法の構築を検討している。この方法が確立できれば、初期段階のリガンド候補絞り込みを短期間で行うことができると考えている。
謝辞
本研究の遂行にあたり、吉川寛非常勤顧問(BRH)、谷村禎一准教授(九大)、Frederic Marion-Poll教授(INRA)、小寺正明助教(京大)、武藤愛研究員(京大)に多大な協力と助言を頂いた。
引用文献
- 1 Thompson, J. N. Evolutionary genetics of oviposition preference in swallowtail butterflies. Evolution 42, 1223-1234 (1988).
- 2 Ohsugi, T., Nishida, R. & Fukami, H. Multi-component system of oviposition stimulants for a Rutaceae-feeding swallowtail butterfly, Papilio xuthus. Applied Entomology and Zoology 26, 29-40 (1991).
- 3 Feeny, P. in Swallowtail Butterflies: Their Ecology and Evolutionary Biology (eds JM Scriber, Y. Tsubaki, & RC. Lederhouse) 9-15 (Scientific Publishers, 1995).
- 4 Tanimura, T., Isono, K., Takamura, T. & Shimada, I. Genetic dimorphism in the taste sensitivity to trehalose in Drosophila melanogaster. J. Comp. Physiol. 141, 433-437 (1982).
- 5 Clyne, P. J., Warr, C. G. & Carlson, J. R. Candidate taste receptors in Drosophila. Science 287, 1830-1834, doi:8337 [pii] (2000).
- 6 Jones, W. D., Cayirlioglu, P., Kadow, I. G. & Vosshall, L. B. Two chemosensory receptors together mediate carbon dioxide detection in Drosophila. Nature 445, 86-90 (2007).
- 7 Chyb, S., Dahanukar, A., Wickens, A. & Carlson, J. R. Drosophila Gr5a encodes a taste receptor tuned to trehalose. Proc Natl Acad Sci U S A 100 Suppl 2, 14526-14530 (2003).
- 8 Galindo, K. & Smith, D. P. A large family of divergent Drosophila odorant-binding proteins expressed in gustatory and olfactory sensilla. Genetics 159, 1059-1072 (2001).
- 9 Matsuo, T., Sugaya, S., Yasukawa, J., Aigaki, T. & Fuyama, Y. Odorant-binding proteins OBP57d and OBP57e affect taste perception and host-plant preference in Drosophila sechellia. PLoS Biol 5, e118, doi:06-PLBI-RA-0167R3 [pii] 10.1371/journal.pbio.0050118 (2007).
- 10 Ozaki, K., Utoguchi, A., Yamada, A. & Yoshikawa, H. Identification and genomic structure of chemosensory proteins (CSP) and odorant binding proteins (OBP) genes expressed in foreleg tarsi of the swallowtail butterfly Papilio xuthus. Insect Biochem Mol Biol 38, 969-976, doi:S0965-1748(08)00133-1 [pii] 10.1016/j.ibmb.2008.07.010 (2008).
- 11 Ozaki, K. et al. A gustatory receptor involved in host-plant recognition for oviposition of the butterfly, Papilio xuthus. Nature communications 2:542 (2011).
- 12 Ryuda, M. et al. Gustatory sensing mechanism coding for multiple oviposition stimulants in the swallowtail butterfly, papilio xuthus. J Neurosci 33, 914-924, doi:10.1523/JNEUROSCI.1405-12.2013 33/3/914 [pii] (2013).
- 13 Grabherr, M. G. et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nat Biotechnol 29, 644-652, doi:10.1038/nbt.1883 nbt.1883 [pii] (2011).
- 14 Murata, T. Mori, N. Nishida, R. Larval feeding stimulants for a rutaceae-feeding swallowtail butterfly, Papilio xuthus L. in Citrus unshiu leaves. J Chem Ecol. 2011 Oct;37(10):1099-109. doi: 10.1007/s10886-011-0022-5.
